ÁREA
Química de Produtos Naturais
Autores
dos Santos Parrião, F.W. (UEA) ; Castro dos Santos, G. (UEA) ; Henrique Ferreira Koolen, H. (UEA) ; Venancia Faria, J. (UEA)
RESUMO
Fungos do gênero Trichoderma são conhecidos por produzirem inúmeras biomoléculas com amplo espectro de atividades biológicas, dentre as quais destacamos aqui a classe dos peptaibols que apresentam efeito antibiótico e antitumoral. Este trabalho discutiu a tentativa de desreplicação de peptaibols obtidos de extratos fúngicos de Trichoderma por meio da plataforma GNPS. Além disso, descreve a anotação manual a partir dos dados HPLC-MS2 de peptaibols oriundos de extratos fúngicos de Trichoderma amazonicum de códigos: MMSRG38A e MMSRG38B, isolados da casca do açaizeiro (Euterpe precatória). No total foram propostas sete sequências putativas de peptaibols, que servirão para pesquisas futuras.
Palavras Chaves
Trichoderma amazonicum; peptaibols; elucidação
Introdução
Estudos das biomoléculas produzidas por linhagens de Trichoderma spp. permitiram a identificação de metabólitos secundários de diferentes classes, tais como alcalóides, fenóis, pironas, terpenóides, esteróides, gliotoxinas, peptídeos, entre outros, responsáveis por um amplo espectro de atividades biológicas (HOU et al, 2022). Almeja-se destacar aqui uma classe específica de peptídeos, chamados peptaibols. Tratam-se de peptídeos não ribossomais isolados, principalmente de fungos, e conhecidos pelo uso como agentes de biocontrole no combate de outros microrganismos fitopatogênicos. Além disso, o Trichoderma ainda é capaz de auxiliar na promoção de crescimento vegetal devido a capacidade de promover fixação de nitrogênio, absorção de fósforo e enxofre, o que auxilia no crescimento e desenvolvimento das plantas (MEYER, MAZARO e SILVA, 2019). A origem da palavra peptaibol deriva da junção de: peptídeo/Aib/ol, a qual está diretamente relacionada à estrutura básica dessa classe de peptídeos. Estes são considerados anfipáticos por conter os seguintes resíduos de aminoácidos apolares em sua estrutura: α- aminoisobutírico (Aib) e a isovalina (Iva) (DANIEL e RODRIGUES FILHO, 2007). Este trabalho visa discutir a elucidação de metabólitos secundários da classe dos peptaibols produzidosTrichoderma amazonicum considerando o grande potencial destes metabólitos secundários para às indústrias agrícola e farmacêutica. O Objetivo é realizar a desreplicação de peptaibols com base análise de dados espectrométricos obtidos via HPLC-MS2 e da rede molecular gerada na plataforma GNPS para os extratos fúngicos de T. amazonicum.
Material e métodos
A metodologia geral até obtenção dos arquivos (raw) analisados no HPLC- MS2 foi feita com base na método descrito por Castro et al, (2023). Pesou-se 2 mg de cada extrato bruto de T. amazonicum provenientes do açaizeiro: amostras MMSG38A (casca interna) e MMSRG38B (casca externa), as quais foram enviadas para análises via Espectrometria de Massas de Alta Resolução. A elucidação manual das sequências de aminoácidos dos peptaibols considerou obtenção dos espectros (MS) que tinham o padrão monoisotópico característico de peptídeos para em seguida avaliar as fragmentações dos espectros (MS/MS) usando o software X-Calibur. A montagem das moléculas foi feita no ChemDraw 12.0 e os cálculos de erro consideraram a massa teórica do padrão monoisotópico dos íons usando o site ChemCalc (http://www.chemcalc.org). Além disso, à desreplicação os espectros de íons produtos oriundos da análise de LC-MS2 dos extratos de T. amazonicum foram analisados e organizados em redes moleculares usando a plataforma GNPS disponível em (http://gnps.ucsd.edu). Os parâmetros utilizados para a inserção dos dados HPLC- MS2 no GNPS consistiram em determinar: a tolerância de massa de íon precursor no valor de 0.05 Da, a tolerância de íon de produto foi de 0.1 Da, o cosseno foi de 0.65 mínimo de seis íons para fragmentos correspondentes limite de 10 nós vizinhos por nodo e mínimo de dois nodos por cluster (WANG et al, 2016).
Resultado e discussão
Em nossa avaliação exploratória do perfil químico, utilizando LC-MS2,
no modo positivo, para o extrato bruto da linhagem T. amazonicum
considerou-se os compostos alvo que apresentaram massa molecular superior a 900
u, como esperado para moléculas de natureza peptídica. Para tal, foi necessário
efetuar uma varredura manual para obtenção dos espectros de fragmentação MS/MS,
onde inicialmente buscou-se por espectros MS que continham padrão monoisotópico
dos íons. A partir disso, foi possível propor a composição dos resíduos de
aminoácidos com base em cada espectro de fragmentação MS/MS. As elucidações
putativas foram feitas manualmente a partir dos fragmentos moleculares propostos
ao considerar as diferenças entre os picos de m/z encontrados nos
espectros MS/MS, cujas massas foram associadas aos resíduos de aminoácidos
propostos. Nesse sentido, destacam-se os dados de fórmula molecular, erro e
tempo de retenção (TR) dos peptaibols propostos: inicialmente peptaibols de 10
resíduos, cujos íons foram: m/z 991.65 (C49H86N10O11, com erro 5,62 ppm,
TR 11,06); m/z 991,64 (C49H86N10O11,-4,45ppm, TR 10,89); m/z
1127,33 (C49H86N14O16, 2,17 ppm, TR 11,05); m/z 1049.64 (C49H84N12O13,
0,95 ppm, TR 11,12); m/z 1005,66 (C50H88N10O11, 1,22 ppm, TR 10,88), um
peptaibol de 11 resíduos m/z (1244,76 (C60H101N13O15, 1,47 ppm, TR 10,98)
e por fim, um peptaibol de 15 resíduos m/z 1481,91 (C66H116N18O20, erro
de massa 6,74 ppm, TR 11,62). A desreplicação não teve êxito, pois as redes
moleculares não foram semelhantes com moléculas já descritas/elucidadas.
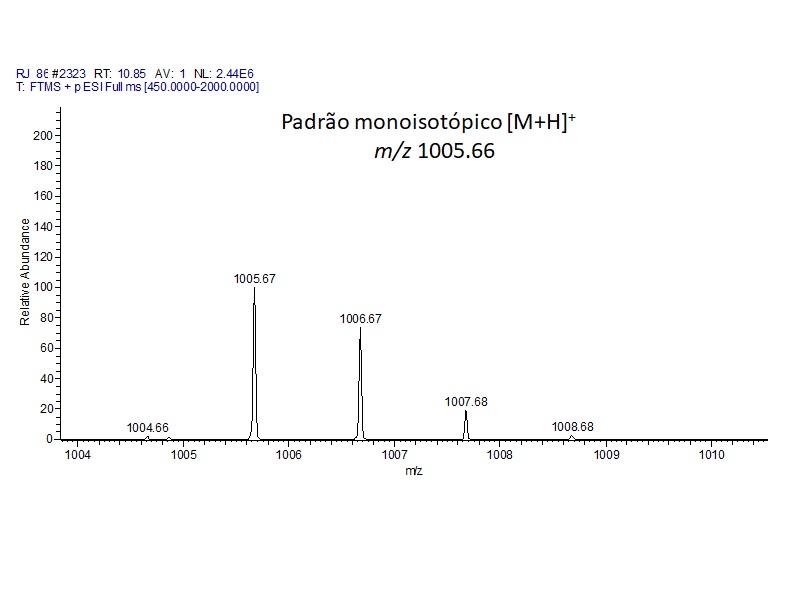
Espectro de massa da molécula protonada [M+H]+
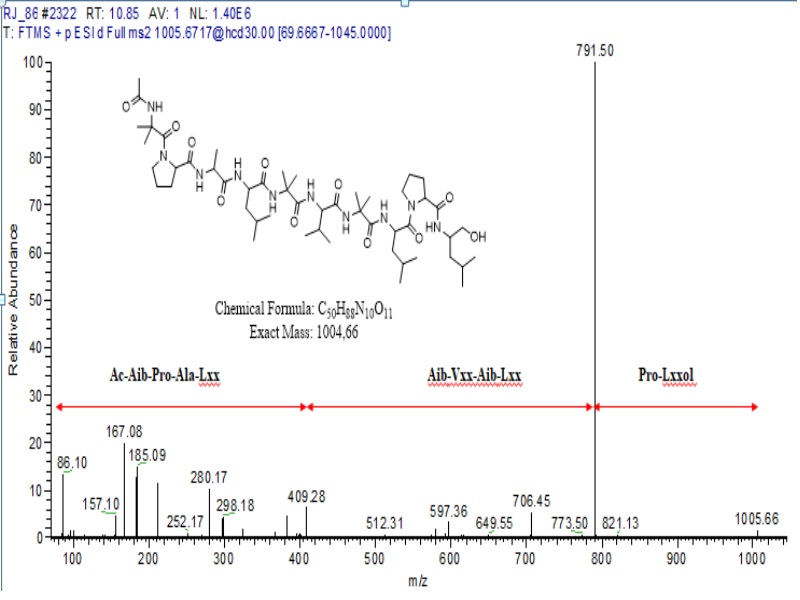
Espectro de MS/MS do íon m/z 1005,66 e sequência \r\nputativa de resíduos do peptaibol.\r\n
Conclusões
Foi possível analisar os dois extratos fúngicos da linhagem T. amazonicum. A partir disso, sete estruturas putativas de peptaibols foram descritas na amostra MMSGR38A. À elucidação manual dos peptaibols empregou-se o software X-calibur e as fragmentações observadas nos espectros HPLC-MS/MS. Notou-se que a ausência de estruturas de peptaibols nas análises desreplicatórias via GNPS não foi definitiva para a determinação da composição dos extratos, em contrapartida, foi possível propor peptaibols de 10, 11 e 15 resíduos.
Agradecimentos
Agradecemos a Universidade do Estado do Amazonas UEA/CEST pelo suporte e a Fundação de Amparo à Pesquisa do Estado do Amazonas (FAPEAM) pelo apoio financeiro edital 001/2021 Mulheres na Ciência.
Referências
CASTRO, G.; SOUSA, T.; da SILVA, G.; PEDROSO, R. C. N.; MENEZES, K. S.; SOARES, M. A.; DIAS, G. M.; SANTOS, A. O.; YAMAGISHI, M. E. B.; FARIA, J. V.; JANUÁRIO, A. H.; KOOLEN, H. H. F. 2023. Characterization of Peptaibols Produced by a Marine Strain of the Fungus Trichoderma endophyticum via Mass Spectrometry, Genome Mining and Phylogeny Based Prediction. Metabolites, v. 13, n. 221, p. 01 17, 2023.
DANIEL, J. F. S.; RODRIGUES FILHO, E. Peptaibols of Trichoderma. Natural Product Reports, v.24, n. 5, p. 1128-1141, 2007.
HOU, X.; SUN, R.; FENG, Y.; ZHANG, R.; ZHU, T.; CHE, Q.; ZHANG, G.; LI, D. Peptaibols: Diversity, biactivity and biosynthesis. Engineering Microbiology, v. 2, p. 10026, 2022.
MEYER, M.; MAZARO, S.; SILVA, J. C. da. Trichoderma: uso na agricultura. 1ª ed., Brasília - DF, EMBRAPA, 2019.
WANG, M. et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology, v. 34, n. 8, p. 828-837, 2016.