ÁREA
Química Medicinal
Autores
Barros Silva, L. (UFPA) ; de Sousa Barros, E. (UNIFAP) ; Viegas da Silva, A. (UNIFAP) ; Patricia Santos de Oliveira, L. (UNIFAP) ; Mendes Nogueira Cardoso, F. (UNIFAP) ; Victor Ferreira dos Santos, I. (UNIFAP) ; da Silva Ramos, R. (UNIFAP) ; Souza Costa, N. (UNIFAP) ; Laurindo Dantas dos Santos Andrad, M. (UNIFAP) ; Aparecido Proietti Junior, A. (UNIFAP) ; Breno Rodrigues dos Santos, C. (UNIFAP)
RESUMO
As substâncias antimicrobianas são componentes químicos capazes de inibir o crescimento ou eliminar microorganismos, como bactérias. O alvo molecular da vancomicina é a parede celular bacteriana. Neste trabalho serão planejados potenciais compostos com atividade antibacteriana com o auxílio de ferramentas computacionais. A atividade antibacteriana será avaliada via Triagem Virtual baseado em ligante por meio da técnica de HCA, bem como na determinação das propriedades farmacocinéticas e toxicológicas (ADME/TOX). Por fim, o método de validação farmacofórica, predição adme/tox, afinidade e energia de ligação mostraram resultados satisfatórios para a predição atividade antibacteriana para VRSA nos dois compostos (LMQC 2 e LMQC3) obtidos via triagem virtual baseado em ligante.
Palavras Chaves
Atividade antibacteriana; VRSA; Triagem virtual
Introdução
Desde os primórdios da civilização, a busca pelo tratamento das principais doenças que acometem a humanidade tem sido uma preocupação constante, especialmente no que diz respeito à atividade antibacteriana e antimicrobiana (Ambrose et al., 2023) O mecanismo de resistência nas cepas Staphylococcus aureus resistente à vancomicina (VRSA) se dá pela existência de um espessamento importante da parede celular bacteriana do S. aureus, que dificulta a penetração dos glicopeptídeos (Vancomicina). A maior parte (>90%) das infecções por VRSA são infecções frequentes na pele e estruturas moles, podendo atingir o sistema nervoso central, o trato respiratório, o trato urinário, causar osteomielite e síndrome do choque tóxico (Assis et al., 2017) Neste estudo, a metodologia de triagem virtual e estratégias in silico será utilizada para descoberta de potenciais compostos terapêuticos que podem ser eficazes contra S. aureus resistente à vancomicina e seletividade MRSA, para resultar na identificação e caracterização de novos alvos de fármacos seguindo a potencialidade do alvo. Ao término do estudo, os desfechos obtidos serão empregados para avaliar os compostos mais promissores que possuam atividade antibacteriana, com seletividade em relação a MRSA e VRSA, demonstrarem potencial atividade antibacteriana e podem ser considerados como candidatos viáveis para investigações futuras em ensaios biológicos e clínicos.
Material e métodos
Uma série de 17 compostos e suas bioatividades antimicrobiana para VRSA coletados da literatura (Casero et al., 2015; Gatadi et al., 2019). Os compostos possuem valores de MIC (concentração inibitória mínima) variando de 0.25 a 2×105 µM. A Tetraciclina será usada como template (composto 01). A geração do modelo farmacofórico realizada no servidor web PharmaGist (http://bioinfo3d.cs.tau.ac.il/pharma/), e a pontuação mais elevada será utilizada para a validação utilizando HCA. Essa validação utilizará os descritores farmacofóricos moleculares obtidos pelo PharmaGist. A validação do modelo será conduzida no software MiniTab (Ferreira et al., 2019). Em seguida, o modelo farmacofórico validado será submetido a uma triagem virtual no servidor on-line Pharmit® (http://pharmit.csb.pitt.edu). Posteriormente, refinado usando o Discovery Studio 4.023 (DS)(Version, 2017), mantendo as coordenadas espaciais (x, y, z e R). Além disso, realizado predições farmacocinéticas e toxicológicas (ADME/Tox) no programa DS. Esse processo abrangente permite a avaliação mais detalhada das propriedades farmacocinéticas e toxicológicas das estruturas selecionadas, além de proporcionar uma base sólida para a tomada de decisões na busca de potenciais candidatos a fármacos(Ramos et al., 2022). O software AutoDock 4.2® (versão 4.2.6, San Diego, Califórnia, CA, EUA) foi usado para a simulação de docking. Os dados das estruturas cristalinas de raios- X da Estrutura de cristal da timidilato quinase de Staphylococcus aureus, ligada ao inibidor, será obtido do Protein Data Bank (PDB ID: 4GSY), e o PDB 4CJN será utilizado para dualidade à MRSA. Todos os procedimentos foram realizados seguindo protocolo de (Silva et al., 2023).
Resultado e discussão
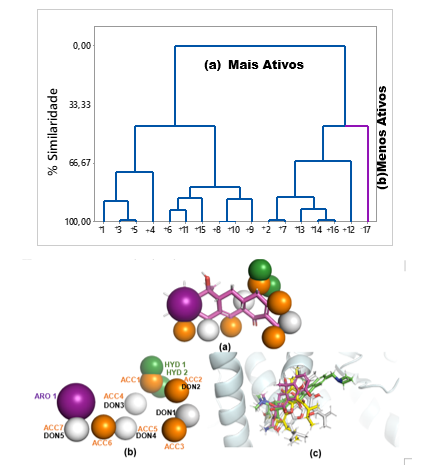
A técnica HCA possibilitou confirmar as duas categorias baseado no valor de pMIC
= logMIC, sendo a variável dependente (pMIC) e independente os descritores
farmacofóricos moleculares [átomos (ATM), Recursos (FEAT), características
espaciais (SF), aromáticos (ARO), hidrofóbicos (HYD), doador de hidrogênio
(DON), aceitador de ligações de hidrogênio (ACC)], mostrado no dendograma de
similaridade estrutural. O modelo farmacofórico escolhido apresentou uma
pontuação de 58,191 com 4 características farmacofóricas: aromática (ARO),
Doador de hidrogênio (DON), hidrofóbica (HYD) e aceitador de ligação de
hidrogênio (ACC), presente no sítio ativo de 4GSY com treze resíduos de
aminoácidos que são Glu-37A, Gly-44A, Arg-48A, Val-51A, Leu-52A, Leu-65A, Phe-
66A, Ser-69A, Arg-70A, Arg- 92A, Ser-97A, Tyr-100A, Gln-101A que são cruciais
para atividade biológica, ver Figura 1.
Utilizando a técnica de triagem virtual, foram identificadas inicialmente 200
estruturas promissoras, seguido por previsões ADME / Tox e inspeção visual.
O docking molecular mostrou um valor de afinidade de ligação (∆G) de -9,30
kcal/mol para a molécula controle. Assim, nossos resultados apresentam um
desempenho satisfatório na previsão da conformação da interação. A análise das
afinidades de ligação aqui calculadas para VRSA indicou que LMQC1, LMQC2 e LMQC3
apresentaram bons resultados quando comparado à molécula 1 (Controle), sendo o
composto LMQC1 apresentou valor de afinidade de ligação (∆G) de -9,50 kcal/mol,
um candidato promissor para potencial na atividade antibacteriana para VRSA, ver
Figura 2.
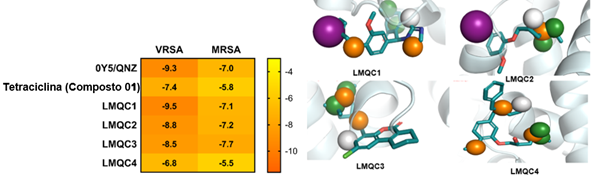
resultantes da triagem virtual do modelo para VRSA \r\n(PDB ID: 4GSY) e MRSA (PDB ID= 4CJN); Tetraciclina -\r\nTemplate. E Estruturas promissoras selecionadas
Conclusões
O HCA demonstrou ser uma ferramenta adequada, uma vez que possibilita a visualização da disposição das moléculas com base em suas similaridades estruturais. Os resultados farmacocinéticos mostram que os quatro compostos possuem bom perfil farmacocinético e toxicológico. Assim, nossos resultados apresentam um desempenho satisfatório na previsão da conformação da interação (afinidade de ligação de -9,30 kcal/mol). É importante destacar que é fundamental submetê-las a testes in vitro ou in vivo para validar a atividade antibacteriana em relação à VRSA para confirmar a atividade in silico.
Agradecimentos
O presente trabalho foi realizado com apoio da Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - Brasil (CAPES) - 01 -88887.645141/2021-00 e aos membros do Laboratório de Modelagem e Química Computacional (LMQC).
Referências
AMBROSE, S. J. et al. Characterisation of an early South African multiply antibiotic-resistant global clone 1 Acinetobacter baumannii isolate. Journal of Global Antimicrobial Resistance, v. 33, p. 337-344, 2023. ISSN 2213-7165.
ASSIS, L. M.; NEDELJKOVIĆ, M.; DESSEN, A. New strategies for targeting and treatment of multi-drug resistant Staphylococcus aureus. Drug Resistance Updates, v. 31, p. 1-14, 2017. ISSN 1368-7646.
CASERO, C. et al. Structure and Antimicrobial Activity of Phloroglucinol Derivatives from Achyrocline satureioides. Journal of natural products (Washington, D.C.), v. 78, n. 1, p. 93-102, 2015. ISSN 0163-3864.
FERREIRA, E. F. et al. Identification of New Inhibitors with Potential Antitumor Activity from Polypeptide Structures via Hierarchical Virtual Screening. Molecules, v. 24, n. 16, p. 2943, 2019.
GATADI, S. et al. Synthesis and evaluation of new quinazolin-4(3H)-one derivatives as potent antibacterial agents against multidrug resistant Staphylococcus aureus and Mycobacterium tuberculosis. European journal of medicinal chemistry, v. 175, p. 287-308, 2019. ISSN 0223-5234.
RAMOS, R. S. et al. Identification of Potential Antiviral Inhibitors from Hydroxychloroquine and 1,2,4,5-Tetraoxanes Analogues and Investigation of the Mechanism of Action in SARS-CoV-2. Int J Mol Sci, v. 23, n. 3, Feb 4 2022. ISSN 1422-0067.
SILVA, L. B. et al. Galantamine Based Novel Acetylcholinesterase Enzyme Inhibitors: A Molecular Modeling Design Approach. Molecules, v. 28, n. 3, p. 1035, 2023. ISSN 1420-3049. Available at: < https://dx.doi.org/10.3390/molecules28031035 >.
VERSION, A. Accelrys Discovery Studio. 4.0. Biovia Inc.: San Diego, CA, USA, 2017.