ÁREA
Química Medicinal
Autores
Viegas da Silva, A. (UNIFAP) ; Heloyse Von Schusterschitz Smith, T. (UNIFAP) ; Victor Ferreira dos Santos, I. (UNIFAP) ; Mendes Nogueira Cardoso, F. (UNIFAP) ; Barros Silva, L. (UNIFAP) ; Patricia Santos de Oliveira, L. (UNIFAP) ; Souza Costa, N. (UNIFAP) ; Laurindo Dantas dos Santos Andrade, M. (UNIFAP) ; da Silva Ramos, R. (UNIFAP) ; Breno Rodrigues dos Santos, C. (UNIFAP)
RESUMO
Staphylococcus aureus é um patógeno oportunista causador de doenças infecciosas, sua resistência a antimicrobianos é um fator preocupante para a saúde mundial. Produtos naturais são alternativas fundamentais como novas antimicrobianos, dentre os quais se encaixam os Flavonoides. O presente estudo visa identificar potenciais compostos flavonoides com atividade antimicrobiana usando Docking Molecular, focando na Proteína PBP2a de S. aureus resistente a meticilina. A triagem virtual resultou em 37 compostos com absorção, penetração sanguínea e solubilidade melhores que o composto pivô Rutin. Foram obtidos 5 compostos com maior afinidade ao sítio ativo da proteína que o composto pivô. O flavonoide Formononetin 7-O- glucoside apresentou a maior energia de afinidade, com valor ΔG= -9,229 kcal/mol
Palavras Chaves
Bioinformática; Triagem virtual ; Staphylococcus aures
Introdução
O Staphylococcus aureus resistente a meticilina (MRSA), uma bactéria Gram- positiva, coloniza preferencialmente a mucosa externa, mas pode se tornar um patógeno oportunista em humanos, desencadeando diversas doenças infecciosas. Sua virulência é atribuída a proteínas de superfície, enzimas e toxinas que causam danos à membrana. A resistência a antimicrobianos faz do S. aureus um destacado agente de infecções hospitalares, sendo prevalente e virulento. A propagação ocorre endogenamente, com 15% dos indivíduos saudáveis hospedando estafilococos na pele e nasofaringe. Disseminação exógena também é possível via contato direto ou objetos contaminados (LIMA et al., 2015). Flavonoides, micronutrientes vegetais, desempenham múltiplas funções, incluindo proteção contra UV, patógenos e atração de polinizadores. Muitos têm propriedades farmacológicas, principalmente atividades anti-inflamatórias e antimicrobianas (SANTOS; RODRIGUES, 2017). Flavonoides são essenciais e diversificados em várias espécies vegetais. A bioinformática é crucial na busca por substâncias terapêuticas. Integra informações biológicas e técnicas computacionais para identificar compostos promissores, otimizando a descoberta de novos agentes. Envolve análise de bases de dados, simulações de interações moleculares, previsão de atividade biológica e triagem virtual (DIAS DE ARAÚJO et al., 2008). Isso economiza tempo e recursos ao selecionar compostos com maior potencial de eficácia. O objetivo deste estudo é identificar novos potenciais compostos com atividade antimicrobiana através do Docking Molecular na Penicillin Binding Protein 2a (PDB2a) de Staphylococcus aureus resistentes a meticilina, sendo um estudo com abordagem da Química Computacional e Bioinformática.
Material e métodos
Para este estudo, foi selecionado o flavonoide Rutin (PUBCHEM ID: 5280805) como composto pivô, no estudo de Masumi, et al. (2022) um conjunto de flavonoides foram acoplados no sítio ativo da PBP2a (PDB: 1MWT 2.45 Å), o composto Rutin obteve uma alta afinidade pelo sítio ativo da proteína, com energia estimada em −9.341 kcal/mol, afinidade maior que a Penicilina G cristalográfica, com energia estimada em −6.35 kcal/mol. Foi feita a triagem virtual de compostos similares ao Rutin no banco de dados PubChem (KIM et al., 2023), usando a ferramenta de “Similaridade Estrutural” e fixando o índice de Tanimoto em 0,9 (90%). A triagem virtual retornou 1000 compostos com a similaridade configurada, os compostos foram baixados em formato .sdf. No software Discovery Studio (BIOVITA FOUNDATION, 2007) foi feito os cálculos dos descritores de ADME (absorção, distribuição, metabolismo e excreção). O critério de inclusão adotado foi o nível de absorção intestinal humana, sendo selecionado para a próxima etapa os compostos que obtiveram os valores de “boa absorção”, “moderada absorção” e “baixa absorção”. Os compostos com “absorção muito baixa” foram excluídos. Assim, de um total de 1000 compostos, apenas 38 compostos se enquadraram nos critérios de inclusão e seguiram para a próxima etapa metodológica. Para a etapa final do estudo, os 38 compostos, mais o composto pivô, tiveram sua estrutura 3D otimizada no método Semi-Empírico com a base de dados AM1 via HyperChem 8 (CHEMPLUS, 2000). Os compostos foram acopladas via Docking Molecular no Web Servidor DockThor (GUEDES et al., 2021) na proteína PBP2a (PDB: 1MWT 2.45 Å) (LIM; STRYNADKA, 2002). A minimização de energia da proteína para o Docking Molecular foi feita via Swiss-PDBViewer 4.1.0 (GUEX; PEITSCH, 1997).
Resultado e discussão
A Figura 1 ilustra os compostos que foram submetidos a previsão farmacocinética,
a triagem mostrou que 38 compostos apresentaram o nível de confiança de 99% para
absorção intestinal humana e penetração no sangue, sendo as elipses as regiões
em que se há a predição de moléculas bem absorvidas, as moléculas que não violam
os parâmetros estabelecidos pelas elipses, possuem maior probabilidade de serem
administrados pela via oral (RAMOS et al., 2021). O composto pivô ficou fora do
intervalo de confiança das elipses, pois apresentou um valor de absorção
intestinal classificado como muito baixo.
37 compostos apresentaram bom valor de solubilidade correspondendo ao intervalo
de -4,1 até -2,0 logSw. Um composto apresentou baixa solubilidade, -6,0 até -4,1
logSw. O composto Rutin apresentou valor de solubilidade de -6,182 logSw sendo
classificado como solubilidade muito baixa (CHENG; MERZ, 2003).
Para o Docking Molecular os resultados variaram da máxima energia de afinidade
de ΔG= -9,229 kcal/mol até a mínima de ΔG= -7,923 kcal/mol. O composto pivô
apresentou uma energia de afinidade de ΔG= -9,077 kcal/mol. A Figura 2 demonstra
os 10 compostos com maior energia de afinidade obtidos e as interações com
resíduos de aminoácidos do composto de maior energia de afinidade, o Flavonoide
Formononetin 7-O-glucoside (PUBCHEM ID: 442813).
Formononetin 7-O-glucoside é uma isoflavona glicosídica derivada da daidzeína, é
um metabólito presente em diversas plantas coníferas e gimnospermas, (LEUNER et
al., 2013). Nos resultados deste estudo o composto mostrou interações do tipo
“Conventional Hydrogen Bond” com os resíduos: Tyr441, Thr444, Tyr519 e Glu602,
além de uma ligação “Pi-Pi Stacked” e “Pi-Alkyl” com os resíduos Tyr446 e
Met641, respectivamente.
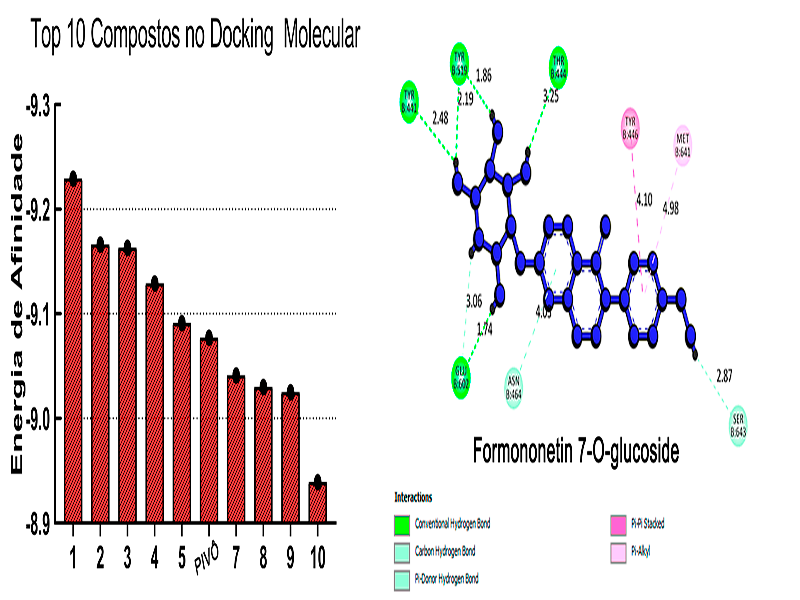
Energia de afinidade dos compostos e pivô e \r\ninterações do Formononetin 7-O-glucoside
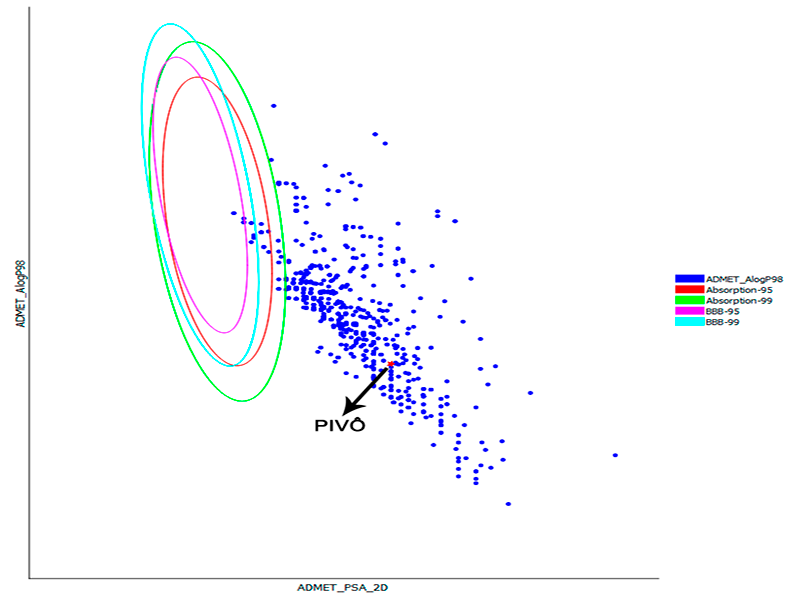
previsão farmacocinética dos 1000 compostos \r\nselecionados mais o Pivô
Conclusões
Neste estudo, dentre 1000 compostos triados a partir do Rutin, 37 exibiram propriedades farmacocinéticas promissoras, não violando o índice de absorção intestinal e penetração sanguínea. Esses compostos mostraram valores farmacocinéticos melhores que o composto pivô. No estudo de Docking Molecular, 5 compostos revelaram maior afinidade ao sítio ativo da Proteína PBP2a do que o composto pivô, sugerindo novas entidades químicas como potenciais inibidores da proteína, o que pode resultar na morte das células bacterianas. As etapas seguintes incluem previsões de toxicidade e simulações de Dinâmica Molecular. Compostos com pontuações mais favoráveis nos estudos in silico serão submetidos a avaliações experimentais para determinar sua atividade antimicrobiana.
Agradecimentos
Agradecimentos ao Laboratório de Modelagem e Química Computacional da Universidade Federal do Amapá pelo empenho, colaboração e orientações científicas para a construção deste trabalho.
Referências
CHEMPLUS. Modular Extensions to HyperChem, Release 8.0, Molecular Modeling for Windows; Hyper, Inc.: Gainesville, FL, USA, 2000.
CHENG, A.; MERZ Jr., K. Prediction of aqueous solubility of a diverse set of compounds using quantitative structure-property relationships. Journal of Medicinal Chemistry, v. 46, p. 3572-3580, 2003.
DIAS DE ARAÚJO, N. et al. [T] A ERA DA BIOINFORMÁTICA: SEU POTENCIAL E SUAS IMPLICAÇÕES PARA AS CIÊNCIAS DA SAÚDE. Disponível em: https://biblat.unam.mx/hevila/Estudosdebiologia/2008/vol30/no70-72/16.pdf. Acesso em: 15 de agosto de 2023.
DO NASCIMENTO CASTILHO, M.; LUIZ VASCONCELLOS VARGAS, A. Tratamentos de infecções por Staphylococcus aureus resistente à meticilina: uma revisão sobre novas possibilidades. Revista de Saúde, [S. l.], v. 14, n. 2, p. 40–54, 2023. DOI: 10.21727/rs.v14i2.3633.
GUEDES, I.A.; BARRETO, A.M.S.; MARINHO, D. et al. New Machine Learning and Physics-Based Scoring Functions for Drug Discovery. Scientific Reports, v. 11, n. 1, p. 3198, 2021. DOI: https://doi.org/10.1038/s41598-021-82410-1.
GUEX, N.; PEITSCH, M. C. SWISS-MODEL and the Swiss-PdbViewer: An environment for comparative protein modeling. Electrophoresis, v. 18, p. 2714-2723, 1997.
KIM, S. et al. PubChem 2023 update. Nucleic Acids Research, v. 51, n. D1, p. D1373-D1380, 2023. DOI: https://doi.org/10.1093/nar/gkac956.
LEUNER, O.; HAVLIK, J.; HUMMELOVA, J.; PROKUDINA, E.; NOVY, P.; KOKOSKA, L. Distribution of isoflavones and coumestrol in neglected tropical and subtropical legumes. Journal of the Science of Food and Agriculture, v. 93, n. 3, p. 575-579, fev. 2013. DOI: 10.1002/jsfa.5835. PMID: 22926873.
LIM, D.; STRYNADKA, N. Structural basis for the β lactam resistance of PBP2a from methicillin-resistant Staphylococcus aureus. Nature Structural & Molecular Biology, v. 9, n. 11, p. 870-876, 2002. DOI: https://doi.org/10.1038/nsb858.
LIMA, M. F. P.; BORGES, M. A.; PARENTE, R. S.; VICTÓRIA JÚNIOR, R. C.; DE OLIVEIRA, M. E. Staphylococcus aureus E AS INFECÇÕES HOSPITALARES – REVISÃO DE LITERATURA. Uningá Review, [S. l.], v. 21, n. 1, 2015.
MASUMI, M; NOORMOHAMMADI, F.; KIANISABA, F.; NOURI, F.; TAHERI, M.; TAHERKHANI, A.; Methicillin-Resistant Staphylococcus aureus: Docking-Based Virtual Screening and Molecular Dynamics Simulations to Identify Potential Penicillin-Binding Protein 2a Inhibitors from Natural Flavonoids. International Journal of Microbiology, v. 2022, Article ID 9130700, 14 pages, 2022. DOI: https://doi.org/10.1155/2022/9130700.
OLIVEIRA, C. F. DE et al. Emergência de Staphylococcus aureus resistentes aos antimicrobianos: um desafio contínuo. Revista de Ciências Médicas e Biológicas, v.13, n. 2 p. 242 – 247, 2014.
RAMOS, R. S. et al. Identification of Potential Antiviral Inhibitors from Hydroxychloroquine and 1,2,4,5-Tetraoxanes Analogues and Investigation of the Mechanism of Action in SARS-CoV-2. International Journal of Molecular Sciences, v. 23, p. 1781, 2022. DOI: https://doi.org/10.3390/ijms23031781.
SANTOS, D. S. DOS; RODRIGUES, M. M. F. Atividades farmacológicas dos flavonoides: um estudo de revisão. Estação Científica, v. 7, n. 3, p. 29–35, 2017.