Autores
Haidar Martorano, L. (UNIVERSIDADE FEDERAL FLUMINENSE) ; Vingre da Silva Mota, G. (UNIVERSIDADE FEDERAL DO PARÁ) ; Ferreira de Albuquerque, A.C. (UNIVERSIDADE FEDERAL FLUMINENSE) ; Alves da Silva, C. (UNIVERSIDADE FEDERAL DE JATAÍ) ; da Silva, A.M. (INSTITUTO FEDERAL CATARINENSE) ; de Jesus Chaves Neto, A.M. (UNIVERSIDADE FEDERAL DO PARÁ) ; Leda Valverde, A. (UNIVERSIDADE FEDERAL FLUMINENSE) ; Paranhos Costa, F.L. (UNIVERSIDADE FEDERAL DE JATAÍ) ; Martins dos Santos Junior, F. (UNIVERSIDADE FEDERAL FLUMINENSE)
Resumo
A combinação de métodos computacionais e Ressonância Magnética Nuclear é uma
ferramenta fundamental na elucidação de novos produtos naturais. Nos últimos anos,
a utilização de procedimentos probabilísticos bayesianos impulsionou o surgimento
de novas abordagens com alto grau de confiança, destacam-se: CP3, DP4 e DP4+. Além
destas, a metodologia ANN-PRA que avalia a correção de uma dada estrutura com base
na qualidade do ajuste obtido pelos valores calculados computacionalmente e
experimentais, por meio da análise do reconhecimento de padrão utilizando redes
neurais artificiais. Neste trabalho, utilizou-se as ferramentas DP4+ e ANN-PRA na
diferenciação do α-(-)bisabolol e seu epímero, (+)-epi-α-bisabolol.
Palavras chaves
Modelagem molecular; Elucidação estrutural; Produtos naturais
Introdução
A elucidação estrutural de novas moléculas não é trivial, ainda mais quando se
trata de produtos naturais complexos, mesmo com a combinação de diversas
técnicas, pode existir grande dificuldade na determinação estrutural. A
combinação da modelagem molecular e dados experimentais de Ressonância Magnética
Nuclear (RMN) revolucionou esta árdua missão (COSTA et al., 2010; COSTA et al.,
2021). No início do século XXI, a maioria dos casos de determinação da
configuração relativa de produtos naturais baseou-se na avaliação da
concordância entre os dados de RMN calculados e experimentais, principalmente,
pelo uso de parâmetros estatísticos simples. Entretanto, para produtos naturais
complexos, esta não é a melhor abordagem, visto que os deslocamentos químicos
simulados para isômeros podem ser muito semelhantes, impossibilitando uma
atribuição inequívoca. Nos últimos anos, a utilização de procedimentos
probabilísticos bayesianos impulsionou o surgimento de novas abordagens com alto
grau de confiança, fomentando uma extensa serie de ferramentas, destacam-se:
CP3, DP4 e DP4+ (COSTA et al., 2021; GRIMBLAT et al., 2015). Outra abordagem de
sucesso, avalia a correção de uma dada estrutura com base na qualidade do ajuste
obtido pelos valores calculados computacionalmente e experimentais, por meio da
análise do reconhecimento de padrões (PRA) utilizando redes neurais artificiais
(RNA), criou-se a metodologia ANN-PRA (ZANARDI e SAROTTI, 2015). Devido ao nosso
continuo interesse na aplicação e desenvolvimento de novas metodologias que
auxiliam a elucidação estrutural de produtos naturais, utilizou-se as
ferramentas DP4+ e ANN-PRA na diferenciação do α-(-)bisabolol e seu epímero,
(+)-epi-α-bisabolol (Fig. 1) (RIGO et al., 2019; ROCHA et al., 2011; ROCHA et
al., 2011).
Material e métodos
Realizou-se uma busca conformacional aleatória para cada candidato, utilizando o
método de Monte Carlo e o campo de força de MMFF no SPARTAN’08 (SPARTAN`08,
Wavefunction inc.). Inicialmente, todas as conformações encontradas dentro de
uma janela de 10kcal.mol-1 foram otimizadas em mPW1PW91/6-31G(d). Os confôrmeros
com energia relativa <2,5kcal.mol-1, que correspondem a mais de 90% da
distribuição de Boltzmann, foram selecionadas para os cálculos de deslocamento
químico de RMN de 1H e 13C. As simulações dos deslocamentos químicos foram
realizadas através da teoria do Funcional da Densidade acoplado ao método GIAO
(GIAO-HDFT), em nível de teoria GIAO/mPW1PW91/6-31G(d)//mPW1PW91/6-31G(d) (COSTA
et al., 2014) utilizando o pacote de software Gaussian 09 (FRISCH et al., 2009).
Os deslocamentos químicos de RMN de 13C e 1H calculados foram escalonados,
respectivamente, através das equações lineares (δesc = 1.05δcalc − 1.22) (COSTA
et al., 2014) e (δesc = 0.98δcalc − 0.09) (CARDOSO et al., 2020). Esta abordagem
diminui os erros sistemáticos relacionados às simulações e aos dados
experimentais. Para a aplicação da ferramenta DP4+, utilizou-se a planilha Excel
fornecida pelos autores. (GRIMBLAT et al., 2015).
Para a aplicação do método ANN-PRA, utilizou-se a planilha Excel fornecida pelos
autores em sarotti-NMR.weebly.com. (ZANARDI e SAROTTI, 2015).
Resultado e discussão
Originalmente, existem 4 estereoisômeros possíveis para o esqueleto proposto,
sendo dois pares de enatiômeros. Como cada par apresenta os mesmos deslocamentos
químicos de RMN em meios isotrópicos, basta calcular um enatiômero de cada
configuração relativa.
Assim, os deslocamentos químicos de RMN para os 2 possíveis estereoisômeros
foram simulados e comparados com os dados obtidos experimentalmente para o
produto natural.
Para aumentar a confiabilidade da discriminação, aplicou-se os deslocamentos
químicos calculados e experimentais na ferramenta DP4+.
A metodologia DP4+ compara os dados experimentais de RMN com os dados simulados
para todas as estruturas possíveis. Através da diferença nos deslocamentos
químicos entre os valores simulados e experimentais e aplicação do teste t de
Student baseado em análise estatística Bayesiana, as estruturas são
classificadas.
A figura 2 mostra os resultados obtidos pelo DP4+ para o αBis e epi-αBis.
Observado a análise C-DP4+, obtida com base nos dados de RMN de 13C, é possível
notar que os resultados individuais são inconclusivos e conflitantes.
Entretanto, em relação aos dados de RMN de 1H (H-DP4+), os resultados obtidos
favorecem a estrutura do αBis em relação ao seu epímero.
Como esperado, a atribuição geral feita por H-DP4+ foi mais significativa que C-
DP4+, resultando em um valor de probabilidade de todos os dados DP4+ de 99,16%
em relação ao candidato indicado pela análise H-DP4+, o isômero αBis.
Para confirmar a atribuição, aplicou-se a metodologia ANN-PRA, que foi criada
com o intuito de minimizar os riscos na elucidação estrutural, uma vez que
classifica a estrutura proposta como correta ou incorreta. No caso deste
trabalho, esta ferramenta concluiu que a estrutura da substância isolada como α-
bisabolol estava correta.
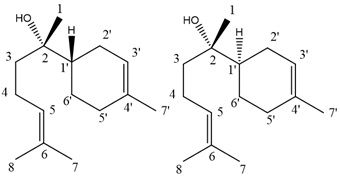
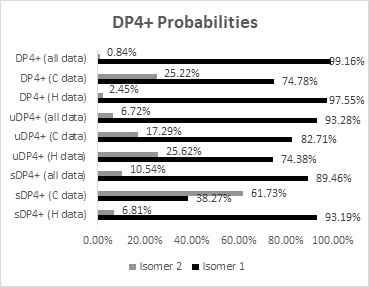
Valores DP4+ obtidos pela correlação dos dados de RMN calculados de αBis (isômero 1) e epi-αBis (isômero 2) com os respectivos dados experimentais.
Conclusões
Neste trabalho, divulgamos e utilizamos as novas ferramentas, como DP4+ e ANN-PRA,
disponíveis para auxiliar o processo de elucidação estrutural de produtos
naturais, através de dados de RMN. Apenas utilizando os dados de RMN de 1H e 13C,
combinados com DP4+ na forma escalonada e não escalonada, foi possível
determinar a configuração relativa da molécula de α-bis, com alto grau de
confiança. Esta proposta foi validada com a aplicação do protocolo ANN-PRA.
Agradecimentos
Agradecimentos: FAPERJ (concessão 211.319-2019), CAPES e CNPq, pelo apoio
financeiro.
Referências
CARDOSO, E.F.; DE ALBUQUERQUE, A.C.F.; NETO, A. M. DE J. C.; MOTA, G.V.S.; COSTA, F.L.P. Gauge-Including-Atomic-Orbitals-mPW1PW91/6-31G(d) Scaling Factor as a Satisfactory Cost-Effectiveness Ratio for H-1 Nuclear Magnetic Resonance Chemical Shift Calculations. Adv. Sci. Eng. Med., 2020, 12, 8.
COSTA, F.L.P.; DE ALBUQUERQUE, A.C.F.; SANTOS JUNIOR, F.M.; AMORIM, M.B. GIAO-HDFT scaling factor for 13C NMR chemical shifts calculations. J. Phys. Org. Chem., 2010, 23, 10.
COSTA, F.L.P.; DE ALBUQUERQUE, A.C.F.; BORGES, R.M.; SANTOS JUNIOR, F.M.; AMORIM, M.B. High cost-effectiveness ratio: GIAO-MPW1PW91/6-31G(d)//MPW1PW91/6-31G(D) scaling factor for 13C nuclear magnetic resonance chemical shifts calculation. J. Comput. Theor. Nanos., 2014, 11,1.
COSTA, F.L.P.; DE ALBUQUERQUE, A.C.F.; FIOROT, R.G.; LIÃO, L.M.; MARTORANO, L.H.; MOTA, G.V.S.; VALVERDE, A.L.; CARNEIRO, J.W.M.; SANTOS JUNIOR, F.M. Structural characterisation of natural products by means of quantum chemical calculations of NMR parameters: new insights. Org. Chem. Front., 2021, 8, 2019.
DOS SANTOS, F. M.; DA SILVA MOTA, G. V.; MARTORANO, L. H.; DE ALBUQUERQUE, A. C. F.; DA SILVA, C. A.; DA SILVA, A. M.; DE JESUS CHAVES NETO, A. M.; VALVERDE, A. L.; CARDOSO, E. F.; COSTA, F. L. P. Combined application of DP4+ and ANN-PRA to determine the relative configuration of natural products: The alpha-bisabol case study. Magn. Reson. Chem. 2022, 60(6), 533.
FIZER, M.; SLIVKA, M.; KOROL, N.; FIZER, O. Identifying and explaining the regioselectivity of alkylation of 1,2,4-triazole-3-thiones using NMR, GIAO and DFT methods. J. Mol. Struct. 2021, 1223, 128973.
FRISCH, M.J. et al., Gaussian 09; Gaussian Inc.: Wallingford, CT, 2009.
GRIMBLAT, N.; ZANARDI, M.M.; SAROTTI, A.M. Beyond DP4: an Improved Probability for the Stereochemical Assignment of Isomeric Compounds using Quantum Chemical Calculations of NMR Shifts. J. Org. Chem., 2015, 80, 12526.
RIGO, A. et al. BID and the α-bisabolol-triggered cell death program: converging on mitochondria and lysosomes. Cell Death Dis., 2019, 10, 12.
ROCHA, N.F.M. et al. (−)-α-Bisabolol-induced gastroprotection is associated with reduction in lipid peroxidation, superoxide dismutase activity and neutrophil migration. Eur. J. Pharm. Sci., 2011, 44, 4.
ROCHA, N.F.M. et al. Anti-nociceptive and anti-inflammatory activities of (−)-α-bisabolol in rodents. Naunyn. Schmiedebergs. Arch. Pharmacol., 2011, 384, 6.
Spartan ’08, Wavefunction Inc., Irvine, CA.
















