Melhoramento da enantiosseletividade de lipases para resolução de álcoois primários aromáticos de interesse farmacêutico: um estudo in silico
- Home
- Trabalhos
ÁREA
Química Medicinal
Autores
Fachini, B.R. (UFPR) ; Maldonado, M.R. (UFPR) ; Alnoch, R.C. (USP) ; Andretta, A.T. (UFPR) ; Krieger, N. (UFPR)
RESUMO
A resolução cinética de racematos através de lipases é usada pela indústria farmacêutica para obter intermediários de compostos bioativos. Como nem todas as lipases possuem boa enantiosseletividade para compostos como álcoois primários aromáticos quirais, o desenho racional de proteínas pode auxiliar no melhoramento dessas propriedades. Este trabalho estudou o mecanismo de resolução dos álcoois pela lipase de metagenômica LipC12, através de docking molecular e desenho racional, a fim de identificar e diminuir a afinidade dos ligantes por cavidades inespecíficas. Após uma série de mutações in silico, análises de afinidade e variação de energia livre de ligação, foi obtida uma variante de LipC12 que apresentou perfis diferentes para os enantiômeros, o que indica maior enantiosseletividade.
Palavras Chaves
Enantiosseletividade; Engenharia de Proteínas; Lipases
Introdução
Lipases (EC. 3.1.1.3) estão entre as enzimas mais aplicadas em química orgânica por apresentarem grande robustez e propriedades de quimio-, régio- e enantiosseletividade, capacidade de aceitar uma variedade de substratos, e catalisarem reações de síntese em solventes orgânicos (DE ALMEIDA et al., 2020). Com o aumento da necessidade de sintetizar compostos enantiomericamente puros, a propriedade enantiosseletiva de lipases é explorada na resolução cinética de racematos e tem sido cada vez mais apreciada. Esses compostos são usados como blocos de construção quiral nas indústrias farmacêutica e agroquímica pois muitas vezes apenas um dos enantiômeros tem atividade biológica e, portanto, biocatalisadores se mostram como uma importante ferramenta para o desenvolvimento desses compostos (CARVALHO et al. 2015). Assim, para melhorar a enantiosseletividade de lipases, a compreensão de como os substratos se comportam em seu túnel catalítico é útil para entender as bases moleculares de sua enantiosseletividade, frequentemente associadas a fatores como impedimento estérico, estabelecimento de ligações de hidrogênio e interações eletrostáticas e de Van der Waals (CHEN et al. 2018). Com essa finalidade, vários autores já estudaram as interações entre enzimas e substratos para buscar aumento da atividade e enantiosseletividade de lipases, ou também para melhorar os resultados do processo como um todo. Um exemplo relevante é o de um estudo que utilizou docking molecular para predizer a enantiosseletividade de algumas lipases e esterases, entre elas Candida antarctica lipase B (CALB), Candida rugosa lipase (CRL), Pseudomonas fluorescens lipase (PFE) e Pseudomonas putida esterase (PPE). Foi utilizado o programa Autodock 4 para realizar o docking entre os substratos quirais e as lipases e esterases para prever a enantiosseletividade com base na energia de docking resultante. No fim do estudo, a predição da enantiopreferência foi bem sucedida em 91% dos pares enzima/substrato (JI; XIAOLING; HONGWEI, 2010). Outro estudo visou a aplicação da resolução cinética de maneira sequencial para obter compostos que servem como blocos de construção quiral para síntese assimétrica de moléculas bioativas, os cicloalcanos benzofundidos. Para isso, os autores utilizaram lipases de diferentes microrganismos, como P. cepacia, C. rugosa e C. antarctica (CALB) e acetato de vinila como doador acila, obtendo-se uma taxa de conversão superior a 80%, que não é desejável. Porém, a enantiosseletividade ainda foi moderada (E = 56) com os substratos selecionados ((R,S)-(2,3-dihidro-1H-inden-1-il)metanol, (R,S)-(1,2,3,4-tetrahidronaftalen-1- il)metanol, (R,S)-(3,4-dihidro-2H-cromen-4-il)metanol) (MARTINS et al., 2015). Uma possível alternativa para a resolução do problema é o desenho racional ou semirracional de enzimas através de técnicas de engenharia de proteínas. Para tanto, o presente trabalho utilizou esses métodos para fazer o melhoramento da enantiosseletividade da lipase de metagenômica LipC12, isolada de solo contaminado por gordura animal. LipC12 foi primeiramente caracterizada por Glogauer et al. (2011), e possui alta atividade contra azeite de oliva (1722 U/mg), comparável a outras lipases comerciais. Porém, seu coeficiente de enantiosseletividade para os substratos (R,S)-2-fenil-1-propanol e (R,S)-2- metil-3-fenil-1-propanol é muito baixo (valores de E ≤ 5). A importância desses substratos está principalmente na sua aplicabilidade na indústria farmacêutica, onde eles podem ser utilizados como intermediários de anti-inflamatórios não esteróides (XIONG et al., 2009) e como precursores de flavonóides, fungicidas e inibidores da biossíntese de colesterol (CONTENTE et al. 2013; MEZZETTI et al., 2005). Assim, um estudo utilizando ferramentas de bioinformática e engenharia de proteínas pode auxiliar na compreensão das bases moleculares responsáveis pela baixa enantiosseletividade para essa classe de compostos, permitindo a obtenção de novas variantes de LipC12 mais enantiosseletivas. Sendo assim, o objetivo deste trabalho é entender o mecanismo da enantiosseletividade de LipC12 para os racematos dos álcoois primários aromáticos, aplicando técnicas de docking molecular para analisar a interação dos substratos com o túnel catalítico da lipase. Após compreender esses mecanismos, serão propostos modelos estruturais de variantes de LipC12 com potencial para resolução dos substratos de interesse com base na análise das variações da energia livre de ligação entre a lipase LipC12 e as suas variantes.
Material e métodos
O modelo estrutural de LipC12 foi obtido através da modelagem por homologia com a lipase de Pseudomonas aeruginosa (pdb ID: 1EX9) através do software Modeller (FISER, A., ŠALI, A., 2003). Foram gerados 500 modelos e o melhor modelo foi selecionado com base no parâmetro DOPE (discrete optimized protein energy), que indica o estado de menor energia e, portanto, mais favorável termodinamicamente. A validação do modelo foi então feita pela geração do diagrama de Ramachandran para verificar se a conformação da lipase assumia parâmetros suficientemente bons, através do software Chimera (PETTERSEN et al., 2004). Para realização do docking molecular, os ligantes (isômeros R e S de 2- fenilpropanol e 2-metil-3-fenilpropanol) foram gerados pelo programa Chimera (PETTERSEN et al., 2004), seguidos por minimização de energia, a fim de gerar a conformação de menor energia com ângulos e distâncias mais favoráveis entre as ligações. Os limites da região para docking molecular foram delineados através da ferramenta Autodock Vina (TROTT et al., 2010) e incluíram todos os resíduos de aminoácidos que compõem o túnel catalítico de LipC12 e variantes. Os átomos de hidrogênio foram adicionados aos modelos de LipC12 e variantes através do AutoDockTools (MORRIS et al., 2009), assim como as cargas de Kollman. A identificação das regiões nas quais havia mais interações com os substratos foi feita com base nas posições propostas em trabalhos prévios utilizando LipC12 (MALDONADO et al., 2021), combinadas aos valores de Delta-G de ligação (kJ mol- 1). Por fim, as análises dos complexos entre lipase e substrato foram feitas com auxílio do programa LigPlot+ (LASKOWSKI et al. 2011), que mostra as interações hidrofóbicas e ligações de hidrogênio entre o ligante e os resíduos de aminoácidos do túnel catalítico da enzima. Os resíduos com maior número de interações e com maior ocorrência nos ligantes testados ((R,S)-2-fenil-1- propanol e (R,S)-2-metil-3-fenil-1-propanol) faziam parte da cavidade do doador acila da lipase, o que poderia levar a um efeito competitivo responsável pelos baixos valores de E para esses compostos. Por isso, esses resíduos foram considerados como posições desejáveis para realização de mutações. As variantes de LipC12 foram então construídas por meio da substituição in silico dos aminoácidos-alvo (V115, L161 e L164), que apresentavam maior prevalência e afinidade pelos substratos, por outros que pudessem resultar em uma melhora na variante. A conformação da cadeia lateral do resíduo mutado foi otimizada manualmente através de uma biblioteca de rotâmeros e o impedimento estérico (sobreposição do raio de Van de Waals) e energias de interações não- covalentes (Energia de Coulomb) foram avaliadas para as diferentes conformações das cadeias laterais das variantes.
Resultado e discussão
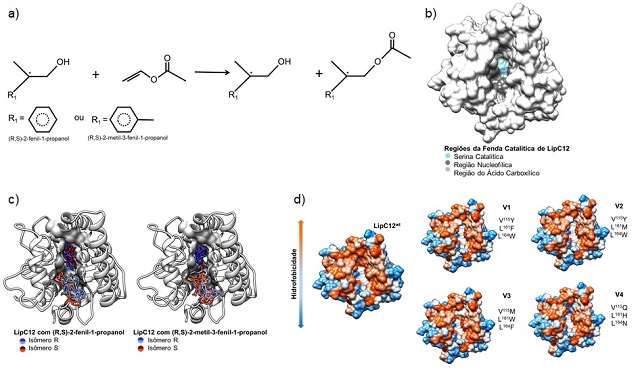
O desenho semirracional de variantes de LipC12 focou na transesterificação dos
álcoois (R,S)-2-fenil-1-propanol e (R,S)-2-metil-3-fenil-1-propanol com acetato
de vinila como doador acila da reação (Figura 1a). Através da realização de
docking molecular observou-se como os ligantes se comportavam no túnel
catalítico da lipase, analisando-se com quais das quatro sub-regiões havia mais
interações: cavidade do oxiânion, cavidades acila, hidrofílica e hidrofóbica. A
cavidade acila é a maior responsável pela enantiosseletividade de ácidos
carboxílicos e as cavidades hidrofílica e hidrofóbica pela de álcoois (Figura
1b). Os resultados de docking molecular de LipC12 sem mutações, aqui denominada
de “selvagem” com a abreviação “wt”, com os substratos mostraram que os
complexos mais estáveis possuem os enantiômeros ligados à cavidade acila, o que
pode indicar competição com o doador acila pela cavidade. Esta preferência pela
cavidade acila foi observada em todos os ligantes analisados (Figura 1c), e este
possível efeito competitivo é indesejável em enzimas para um processo
industrial, uma vez que influencia diretamente as conversões da reação,
tornando-as mais lentas.
A partir dos complexos mais estáveis, foram identificados os resíduos de
aminoácido na cavidade acila de LipC12 que mais interagiam com os álcoois
quirais de interesse. As posições V115, L161 e L164 apareciam com maior
frequência e por isso foram escolhidas para mutações. Visto que a cavidade acila
de LipC12 wt possui hidrofobicidade expressiva (Figura 1d), as mutações
propostas para as variantes desenvolvidas visaram torná-la menos hidrofóbica,
para diminuir sua afinidade pelos álcoois. As mutações realizadas nas variantes
desenhadas, V1, V2, V3 e V4, resultaram na diminuição progressiva da
hidrofobicidade (Figura 1 d).
Figura 1. a) Reação modelo para o desenho racional da lipase LipC12. b) Sítios
responsaveis por enantiosseletividade em LipC12: região em cinza escuro,
enantiosseletividade de álcoois ou nucleófilos quirais e, em cinza claro,
enantiosseletividade de ácidos carboxílicos. c) Complexos de LipC12 wt com os
enantiômeros dos álcoois primários. As estruturas de cor mais escura, na região
do ácido carboxílico possuem menor energia e, portanto, maior afinidade pelo
substrato. d) Hidrofobicidade do túnel catalítico de LipC12 wt e das variantes
V1, V2, V3 e V4, com mutações realizadas na cavidade acila.
Além da análise da hidrofobicidade da cavidade acila e do número de interações
hidrofóbicas e de ligações de hidrogênio, a decisão sobre a melhor variante foi
tomada com base na análise das variações de energia livre de ligação entre a
enzima e o substrato. Essa análise permite um conhecimento maior sobre a
afinidade do composto por uma cavidade específica do túnel catalítico da lipase.
Como o interesse inicial era comparar apenas a cavidade acila com as demais, a
análise energética foi separada apenas em duas partes, descritas abaixo.
Primeiramente, foram avaliados os valores médios de Delta-G de ligação para as
variantes, sendo que V1, V2 e V3 apresentaram os valores mais negativos para a
cavidade acila (-25,73, -25,59 e -25,24 kJ/mol, respectivamente) em relação à
LipC12 wt (Delta-G = -22,38 kJ/mol). Assim, as mutações em V1, V2 e V3
aumentaram a afinidade dos álcoois por essa cavidade, ao passo que as mutações
de V4 levaram a uma diminuição da afinidade quando comparada à enzima selvagem,
pois apresentou valores maiores de Delta-G (-21,27 kJ/mol). Para as demais
cavidades, os valores médios de Delta-G não tiveram alteração expressiva,
sugerindo que as mutações exercem efeitos apenas na cavidade acila (LipC12 wt =
-20,22 kJ/mol, V1 = -20,30 kJ/mol, V2 = -20,40 kJ/mol, V3 = -20,19 kJ/mol e V4=
-20,19 kJ/mol). Outro critério avaliado foi a afinidade do acetato de vinila
pela cavidade acila, mas também não houve variação relevante dos valores de
Delta-G entre LipC12 wt (-14,23 kJ/mol) e V4 (-14,92 kJ/mol), o que sugere que a
ligação do doador acila à cavidade não foi afetada pelas mutações.
Assim, a variante V4, por apresentar uma cavidade acila com menor afinidade
pelos álcoois primários aromáticos quirais, foi usada como base para a etapa
posterior deste trabalho, que consistiu em realizar mutações que pudessem
propiciar o aumento da discriminação entre o par de enantiômeros. A nova
variante derivada de V4, V4.1, possui mutações na cavidade hidrofílica (D20W,
Y26F, P27A, Y28F) e hidrofóbica (M223Y, N265F, Q266A), que levaram a uma
inversão da hidrofobicidade destas regiões (Figura 2 a). Em lipases, a cavidade
hidrofóbica está associada à ligação das partes mais apolares dos substratos, no
caso dos substratos de interesse, essa porção é o anel aromático. Já a cavidade
hidrofílica auxilia no direcionamento do substrato à serina catalítica. A
inversão destas regiões produziu uma alteração no perfil de interação entre os
substratos e V4.1 (Figura 2b), mostrando que, após a etapa de acilação da
enzima, a nova preferência dos álcoois passa a ser a região formada pelos
resíduos W20/F26/A27/F28, que ficam mais orientados à serina catalítica em
comparação aos complexos com LipC12 wt.
Figura 2. a) Comparativo entre a lipase LipC12 wt e variante V4.1, com foco no
túnel catalítico, mostrando as diferenças na hidrofobicidade. b) Comparação
gráfica do perfil de interação entre os álcoois primários e LipC12 wt e entre os
álcoois e V4.1. c) Comparação gráfica do perfil de interação dos isômeros R e S
de 2-fenil-1-propanol com a cavidade acila de LipC12 wt acilada, e com a
cavidade acila de V4.1 acilada.
Além da modificação da região de preferência, V4.1 também aparenta ter uma
melhor discriminação entre os isômeros R e S, uma vez que nos perfis de
interações relativas após a etapa de acilação, o isômero S passa a interagir com
mais resíduos do que o isômero R, o que não é observado em LipC12 wt (Figura 2
c) e representa uma alteração na enantiopreferência da variante V4.1. Comumente,
a discriminação entre enantiômeros tem sido associada à regra de Kazlauskas
(KAZLAUSKAS et al. 1991), que consiste na previsão da enantiosseletividade
através de bolsões de encaixe (bolsão maior e bolsão médio) para os
substituintes ao redor do centro quiral do substrato.
Entretanto, a enantiosseletividade tende a dar-se de forma mais complexa,
envolvendo interações entre a enzima e o substrato, como ligações de hidrogênio,
interações hidrofóbicas e iônicas, e também por impedimento estérico,
particularmente no túnel catalítico (CHEN et al. 2018). A abordagem por docking
molecular foca nesses tipos de interações, podendo revelar o grau de interação
dos isômeros com o túnel catalítico da lipase. Essa diferença no número, tipo e
perfil de interações entre os isômeros pode fazer com que a lipase tenha sua
enantiopreferência aumentada ou diminuída, ressaltando-se que, quanto maior a
discriminação entre os isômeros maior a enantiosseletividade de uma enzima.
Nesse sentido, visando a uma maior diferenciação entre as interações dos
enantiômeros através de ferramentas de engenharia de proteínas, trabalhou-se com
duas hipóteses baseadas na afinidade dos enantiômeros pelo túnel catalítico. Na
primeira hipótese, um dos enantiômeros tem uma afinidade muito maior em
comparação ao outro, o que faz com que ele aja como um inibidor, favorecendo a
conversão do enantiômero de menor afinidade. Já na segunda hipótese, o
enantiômero que tem mais afinidade pelo túnel catalítico é convertido
normalmente, enquanto o outro, que possui menor afinidade, possui dificuldades
para interagir com o túnel catalítico, o que diminui sua velocidade de
conversão. Dentre as hipóteses levantadas, é difícil prever com precisão qual
enantiômero será preferido para a conversão, bem como qual das duas hipóteses é
válida para esse mecanismo. Porém, a diferença visível entre os perfis de
interação observados já pode servir como base para o desenvolvimento de uma
variante de lipase in silico para a resolução de álcoois primários quirais com
maior enantiosseletividade.
A legenda da Figura 1 se encontra nos Resultados e Discussão.

A legenda da Figura 2 se encontra nos Resultados e Discussão.
Conclusões
Através da realização de quatro séries de mutações in silico em três posições- chave (V115, L161 e L164) da cavidade acila da lipase de metagenômica LipC12, foi possível selecionar uma variante com perfil energético favorável, além de modificações no túnel catalítico para diminuição da hidrofobicidade visando à resolução dos álcoois de interesse. Dessa maneira, a variante V4, com mutações V115Q, L161H e L164N, foi selecionada para utilização na próxima etapa do desenho semirracional da de LipC12, que consistiu na engenharia das cavidades hidrofílica e hidrofóbica, para melhorar suas afinidades pelos álcoois primários aromáticos quirais. A variante V4.1, com sete mutações propostas (D20W, Y26F, P27A, Y28F, M223Y, N265F, Q266A), foi desenhada partir das análises dos perfis de interação e, in silico, demonstrou-se que os isômeros R e S dos substratos testados interagem com regiões diferentes do túnel catalítico, o que sugere que esta variante apresenta maior enantiopreferência para um dos isômeros. Além disso, V4.1 também pode apresentar um perfil com menor competição do doador acila pelos substratos. O uso de docking molecular mostrou-se uma técnica útil no desenho semirracional da lipase LipC12 através da identificação de sítios de afinidade, seguidos de mutagênese in silico e análise dos perfis de interação das novas variantes. O presente trabalho foi desenvolvido no âmbito da bolsa PIBIC, fomentada pelo CNPq e pela Fundação Araucária e está vinculado ao Laboratório de Tecnologia Enzimática e Biocatálise (LTEB) do Departamento de Química de Universidade Federal do Paraná (UFPR).
Agradecimentos
A estudante Beatriz R. Fachini recebeu bolsa Fundação Araucária e CNPq, e Marcos R. Maldonado recebeu bolsa CAPES, e Nadia Krieger teve financiamento CNPq (441015/2016-6).
Referências
CARVALHO, A. C. L. D. M., FONSECA T. D. S., DE MATTOS M. C., DE OLIVEIRA M. D. C. F., DE LEMOS T. M. L. G., MOLINARI F., ROMANO D., SERRA I. Recent advances in lipase-mediated preparation of pharmaceuticals and their intermediates, International Journal of Molecular Sciences, 16, 29682–29716, 2015.
CHEN, H., MENG, X., XU, X., LIU, W., LI, S. The molecular basis for lipase stereoselectivity. Applied Microbiology and Biotechnology, vol. 102, issue 8, 3487–3495, 2018.
CONTENTE, M., GRANATO, T., REMELLI, W., ZAMBELLI, P., RAIMONDI, S., ROSSI, M., ROMANO, D. Complementary microbial approaches for the preparation of optically pure aromatic molecules. Annals of Microbiology, 63(3), 1021–1027, 2013.
DE ALMEIDA, D. K. C., DA SILVA, M. R., DE MATTOS, M. C., NUNES, F. M., BALLEREAU, S., GÉNISSON, Y., MARAVAL, V., CHAUVIN, R., OLIVEIRA, M. C. F. Lipase-catalysed enantioselective kinetic resolution of rac-lipidic alkynylcarbinols and a C5 synthon thereof via a hydrolysis approach. Molecular Catalysis, 488, 2020.
FISER, A., ŠALI, A. MODELLER: generation and refinement of homology-based protein structure models. In Methods in Enzymology, C.W. Carter and R.M. Sweet, eds. Academic Press, San Diego, 374, 463-493, 2003.
GLOGAUER, A., MARTINI, V. P., FAORO, H., COUTO, G. H., MÜLLER-SANTOS, M., MONTEIRO, R. A., MITCHELL, D. A., DE SOUZA, E. M., PEDROSA, F. O., KRIEGER, N. Identification and characterization of a new true lipase isolated through metagenomic approach. Microbial Cell Factories, 10, 2011.
JI, L., XIAOLING, T., HONGWEI, Y. Prediction of the enantioselectivity of lipases and esterases by molecular docking method with modified force field parameters. Biotechnology and Bioengineering, 105(4), 687–696, 2010.
KAZLAUSKAS R.J., WEISSFLOCH A.N.E., RAPPAPORT A.T., CUCCIA L.A. A rule to predict which enantiomer of a secondary alcohol reacts faster in reactions catalyzed by cholesterol esterase, lipase from Pseudomonas-cepacia, and lipase from Candida-rugosa. Journal of Organic Chemistry, 56, 2656–2665, 1991.
LASKOWSKI, R. A., SWINDELLS, M. B. LigPlot+: multiple ligand-protein interaction diagrams for drug discovery. Journal of Chemical Information and Modeling, 51, 2778-2786, 2011.
MALDONADO, M. R., ALNOCH, R. C., DE ALMEIDA, J. M., SANTOS, L. A. DOS, ANDRETTA, A. T., ROPAÍN, R. DEL P. C., DE SOUZA, E. M., MITCHELL, D. A., KRIEGER, N. Key mutation sites for improvement of the enantioselectivity of lipases through protein engineering. Biochemical Engineering Journal, 172, 2021.
MARTINS, R. S., AHMAD, A., SILVA, L. F., ANDRADE, L. H. Exploiting sequential lipase-catalyzed reactions to achieve enantiomerically pure chiral primary alcohols. RSC Advances, 5(70), 56599–56605, 2015.
MEZZETTI, A., SCHRAG, J. D., CHEONG, C. S., KAZLAUSKAS, R. J. Mirror-image packing in enantiomer discrimination: Molecular basis for the enantioselectivity of B. cepacia lipase toward 2-methyl-3-phenyl-1-propanol. Chemistry and Biology, 12(4), 427–437, 2005.
PETTERSEN, E.F., GODDARD, T.D., HUANG, C.C., COUCH, G.S., GREENBLATT, D.M., MENG, E.C., FERRIN, T.E. UCSF Chimera - A Visualization System for Exploratory Research and Analysis. Journal of Computational Chemistry, 25(13), 1605-1612, 2004.
TROTT, O., OLSON, A. J. AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry, 31, 455-461, 2010.
XIONG, W. W., WANG, W. F., ZHAO, L., SONG, Q., YUAN, L. M. Chiral separation of (R,S)-2-phenyl-1-propanol through glutaraldehyde-crosslinked chitosan membranes. Journal of Membrane Science, 328(1–2), 268–272, 2009.


