Quantificação de mercúrio em fígado de tambaqui (Colossoma macropomum) da região da Amazônia/Brasil
ISBN 978-85-85905-25-5
Área
Bioquímica e Biotecnologia
Autores
Martins, R.A. (UNIVERSIDADE ESTADUAL PAULISTA) ; Vieira, J.C.S. (UNIVERSIDADE FEDERAL DE MATO GROSSO DO SUL) ; Assunção, A.S.A. (UNIVERSIDADE ESTADUAL PAULISTA) ; de Oliveira, L.C.S. (UNIVERSIDADE FEDERAL DE MATO GROSSO DO SUL) ; de Oliveira, G. (UNIVERSIDADE ESTADUAL PAULISTA) ; Bataglioli, I.C. (UNIVERSIDADE ESTADUAL PAULISTA) ; Padilha, P.M. (UNIVERSIDADE ESTADUAL PAULISTA)
Resumo
Este estudo teve como objetivo quantificar o mercúrio e identificar proteínas biomarcadoras desse metal em amostras de tecido hepático do peixe tambaqui (Colossoma macropomum) da região amazônica brasileira. Para a separação do proteoma hepático utilizou-se a técnica de eletroforese bidimensional em gel de poliacrilamida (2D-PAGE). A quantificação do mercúrio nos spots proteicos foi realizada por espectrometria de absorção atômica em forno de grafite (GFAAS). Os spots proteicos que apresentaram mercúrio foram caracterizados por espectrometria de massa (ESI-MS/MS). As concentrações de mercúrio ficaram na faixa de 0,4018 e 0,4555 ppm. Através da ESI-MS/MS foi possível identificar 40 proteínas, sendo 18 caracterizadas como possíveis biomarcadoras de mercúrio.
Palavras chaves
Biomarcador; GFAAS; Mercúrio
Introdução
O mercúrio (Hg) é um elemento químico responsável por contaminações ambientais devido ao seu alto poder de toxicidade. Este poluente se difunde facilmente no meio ambiente, sendo quimicamente transformado e bioacumulado nos tecidos dos organismos aquáticos (BITTARELLO et al., 2019). A contaminação por mercúrio na Amazônia brasileira ocorreu entre 1980 e meados da década de 1990 em função da intensa atividade de mineração do ouro. Atualmente, a construção e a operação de usinas hidroelétricas na região tem sido motivo de preocupação crescente, em virtude das consequentes mudanças ambientais que podem resultar na remobilização e modificação das espécies químicas de mercúrio, tornando-o disponível para absorção pela biota aquática (QUEIROZ et al., 2018). O consumo de peixes contaminados com mercúrio trazem grandes riscos para a saúde humana, causando disfunções celulares associadas a inativação enzimática, alterações nas conformações das proteínas e consequentemente distúrbio de vários processos metabólicos e de transporte (BITTARELLO et al., 2019). Desta forma, a identificação de biomarcadores proteicos sensíveis e específicos a concentrações de mercúrio em peixes pode auxiliar no monitoramento de seus níveis no ecossistema aquático (VIEIRA et al., 2017). Neste sentido, o objetivo com o presente estudo foi determinar a concentração de mercúrio em amostras de tecido hepático do peixe tambaqui e identificar possíveis biomarcadores associados com a exposição desse metal nos peixes amazônicos.
Material e métodos
Os peixes foram capturados no rio Madeira na região de Porto Velho, Rondônia, Brasil. Para extração da fração proteica, as amostras dos tecidos hepáticos dos peixes foram maceradas na presença de nitrogênio líquido (N2) utilizando um almofariz e pilão. As proteínas obtidas foram precipitadas por meio de misturas com etanol/clorofórmio e etanol/ácido clorídrico. O precipitado foi lavado com etanol gelado e então foi realizada a determinação da concentração de proteína total pelo método de Biureto. Em seguida, os pellets proteicos foram ressolubilizados em tampão específico e submetidos ao processo de separação por eletroforese bidimensional em gel de poliacrilamida (2D-PAGE, gel de 15%). Os spots foram recortados dos géis e mineralizados por processo de digestão ácida (sulfúrico/peróxido de hidrogênio 4:1). A concentração de Hg foi determinada por espectrometria de absorção atômica em forno de grafite (GFAAS) após a mineralização ácida de acordo com os procedimentos descritos por MORAES et al. (2013). Os spots que apresentaram Hg, foram levados para caracterização das proteínas por espectrometria de massa (ESI-MS/MS) para a identificação de possíveis biomarcadores proteicos de Hg.
Resultado e discussão
Foram identificados 9 spots proteicos com a presença de Hg (Tabela 1) e um
total de 40 proteínas distribuídas entre os spots. As concentrações de Hg
encontradas nos spots ficaram entre 0,4018 e 0,4555 ppm. Dentre as proteínas
identificadas, 18 possuem características que favorecem a ligação com o Hg
podendo ser consideradas como potenciais biomarcadoras desse metal, sendo
elas: Fatty acid-binding heart-like protein; Heart-type fatty-acid binding
protein; Keratin; Keratin, type I cytoskeletal 50 kDa-like isoform X3;
Keratin 5; Keratin-like protein; Keratin 8; Keratin, type II cytoskeletal 8
isoform X1; Parvalbumin; Parvalbumin beta; Parvalbumin 4; Superoxide
dismutase [Cu-Zn]; Betaine-homocysteine methyltransferase; Creatine kinase;
Creatine kinase M2-CK; Muscle creatine kinase; Creatine kinase M-type e
Creatine kinase, muscle a. As proteínas fatty acid-binding caracterizadas
nos spots proteicos estão associadas ao transporte e metabolismo de
lipídeos, possuindo em sua estrutura grupos tióis (-SH) que apresentam
grande afinidade com o mercúrio e outros íons metálicos classificados como
“ácidos moles” (BITTARELLO et al., 2019; QUEIROZ et al., 2018). A
parvalbumina e suas isoformas são proteínas envolvidas na ligação de íons
metálicos divalentes como o cálcio, e a sua isoforma beta contém um resíduo
de cisteína que pode formar ligações específicas com o mercúrio,
demonstrando grande potencial para ser utilizada como um biomarcador desse
metal poluente (BRAGA et al., 2015; QUEIROZ et al., 2018). Assim como as
parvalbuminas, a superoxide dismutase [Cu-Zn] e a Betaine-homocysteine
methyltransferase possuem sítios específicos de ligação com o Zn que podem
se ligar a outros íons divalentes como o mercúrio.
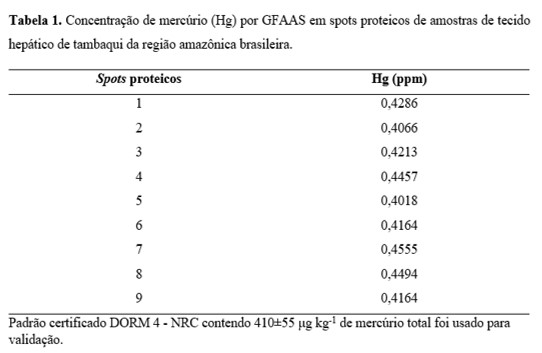
Concentração de mercúrio (Hg) por GFAAS em spots proteicos de amostras de tecido hepático de tambaqui da região amazônica brasileira.
Conclusões
As concentrações de mercúrio em amostras de tecido hepático de tambaqui ficaram entre 0,4018 e 0,4555 ppm. Por meio da ESI-MS/MS foram identificadas 40 proteínas, das quais 18 foram caracterizadas como potenciais candidatas a biomarcadoras de mercúrio.
Agradecimentos
CNPq e FAPESP
Referências
BITTARELLO, A. C.; VIEIRA, J. C. S.; BRAGA C. P.; DE PAULA ARAÚJO, W. L.; BATAGLIOLI, I. C.; DA SILVA, J. M.; BUZALAF, M. A. R.; FLEURI, L. F.; PADILHA, P. M. Characterization of molecular biomarkers of mercury exposure to muscle tissue of Plagioscion squamosissimus and Colossoma macropomum from the Amazon region. Food Chemistry, v. 276, p. 247–254, 2019.
BRAGA, C. P.; BITTARELLO, A. C.; PADILHA C. C. F.; LEITE, A. L.; MORAES, P. M.; BUZALAF, M. A. R.; ZARA, L. F.; PADILHA, P. M. Mercury fractionation in dourada (Brachyplatystoma rousseauxii) of the Madeira River in Brazil using metalloproteomic strategies. Talanta, v. 132, p. 239–244, 2015.
MORAES, P. M.; SANTOS, F. A.; CAVECCI, B.; PADILHA, C. C. F.; VIEIRA, J. C. S.; ROLDAN, P. S.; PADILHA, P. M. GFAAS determination of mercury in muscle samples of fish from Amazon, Brazil. Food Chemistry., v. 141, p. 2614-2617, 2013.
QUEIROZ, J. V.; VIEIRA, J. C. S.; DE OLIVEIRA, G.; BRAGA, C. P.; BATAGLIOLI, I. C.; DA SILVA, J. M.; DE PAULA ARAÚJO, W. L.; PADILHA, P. M. Identification of Biomarkers of Mercury Contamination in Brachyplatystoma filamentosum of the Madeira River, Brazil, Using Metalloproteomic Strategies. Biological Trace Element Research, v. 183, p. 1-10, 2018.
VIEIRA, J. C. S.; BRAGA, C. P.; DE OLIVEIRA, G.; DE LIMA, A. L.; QUEIROZ, J. V.; CAVECCI, B.; BITTARELLO, A. C.; BUZALAF, M. A. R.; ZARA, L. F.; PADILHA, P.M. Identification of protein biomarkers of mercury toxicity in fish. Environmental Chemistry Letters, p. 717-724, 2017.









