Predição estrutural da enzima Nitrate reductase de Solanum lycopersicum (tomate) por homologia molecular.
ISBN 978-85-85905-21-7
Área
Bioquímica e Biotecnologia
Autores
Souza, F.B.S. (UFPA) ; Souza, B.A.S. (UFPA) ; Rodrigues, C.C. (UFPA) ; Siqueira, L.M.M. (UFPA) ; Conceição, G.S. (UFPA) ; Magalhães, V.A. (UFPA) ; Santos, M.L. (UFPA) ; Brasil, D.S.B. (UFPA)
Resumo
O tomateiro (Solanum lycopersicum) é uma das principais cultivares do Brasil, maior produtor desta hortaliça na América do Sul. Nas raízes desta espécie, encontra-se a enzima Nitrate reductase responsável pela produção do metabólito nitrato a partir da conversão catalítica do nitrito. Este produto possui várias aplicações tecnológicas em diversos setores como o farmacêutico, eletroquímico, fertilizantes e biotecnológico. O modelo foi construído a partir da estrutura cristalográfica da enzima de código 1CNF depositada no banco de dados Protein Data Bank (PDB). A validação foi realizada pelo software Pymol com o valor de RMSD igual a 0,097Å e o servidor PSVS com o gráfico de Ramachandran com 90,6% dos resíduos em regiões favoráveis.
Palavras chaves
Predição Estrutural; Nitrate reductase; Homologia
Introdução
O tomate pertence ao gênero Solanum (Solanaceae) e é originário da região dos Andes, adapta-se facilmente a diferentes condições climáticas, sendo este o principal fator de seu amplo cultivo ao redor do mundo (CHETELAT et al., 2009). O cultivo do tomate para processamento industrial ocorre desde o começo do século XX e encontra-se bem estabelecido nas regiões Sudeste, Nordeste e Centro-Oeste, o Brasil é o seu maior produtor na América do Sul (EMBRAPA, 2006). A enzima Nitrate reductase (N. reductase) presente nas raízes do tomateiro é importante para o desenvolvimento destas estruturas responsáveis pela captura de nutrientes, especialmente o ferro. A N. reductase está envolvida no primeiro passo da assimilação de nitratos em plantas, através da conversão de nitrito (NO-2) em nitrato (NO-3) na presença do cofator NAD. O nitrato possui várias aplicações tecnológicas tais como a produção de fertilizantes químicos nitrogenados (KAIWEN et al., 2015), sensor eletroquímico (CARON et al., 2016) e elaboração de fármacos (GUIMARÃES et al., 1999). O objetivo deste trabalho é a elucidação da estrutura tridimensional da N. reductase que permitirá a compreensão do seu mecanismo catalítico enzimático, abrindo um horizonte de possibilidades para o desenvolvimento de novos produtos. A modelagem por homologia molecular consiste em um conjunto de ferramentas para a construção, edição e visualização de sistemas moleculares complexos (Barreiros et al., 1997).
Material e métodos
A predição estrutural da proteína N. reductase foi realizada utilizando o servidor UniProt para a obtenção da sequência de aminoácidos. O alinhamento das sequências foi realizado no servidor Protein Data Bank (PDB) para encontrar os moldes (templates) que estão diretamente relacionados ao alvo (target), através dos parâmetros identidade e similaridade. No software Easy Modeller, efetuou-se o alinhamento entre o alvo e o melhor molde escolhido e posterior construção do modelo tridimensional. A validação do modelo foi realizada no servidor PSVS e software Pymol.
Resultado e discussão
O modelo tridimensional obtido após a realização da predição estrutural da
enzima N. reductase é mostrado na figura 1, o RMSD de valor igual a 0,097Å,
corresponde ao desvio entre a posição do molde cristalográfico de código pdb
1CNF, em vermelho, e o alvo, em verde.O gráfico de Ramachandran- figura 2-
pelo método de Procheck foi construído a fim de avaliar a estereoquímica do
modelo. O número de aminoácidos localizados em regiões com ângulos
favoráveis é dado em porcentagem, o valor para o modelo gerado corresponde a
90,6% dos resíduos em regiões favoráveis, sendo considerado satisfatório
valores acima de 90% conforme Laskowski e colaboradores, em 1993.
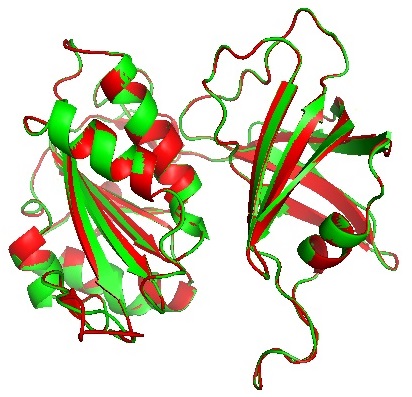
Sobreposição entre o modelo gerado e o molde cristalográfico.
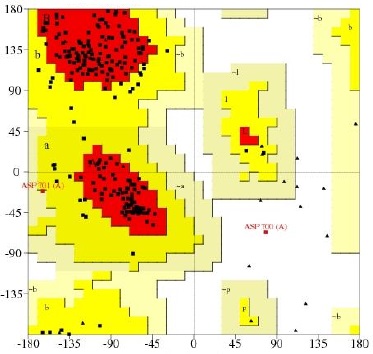
Gráfico de Ramachandran.
Conclusões
O presente modelo encontra-se em processo final de validação para posteriormente ser depositado no banco de dados PDB, através da construção do modelo tridimensional da proteína N. reductase será possível realizar estudos de ancoragem e dinâmica molecular permitindo o estudo das interações moleculares entre esta proteína e outros ligantes.
Agradecimentos
Ao Laboratório de Simulação Computacional, do Departamento de Engenharia Química, da Universidade Federal do Pará.
Referências
CARON, W.; LAMHAMEDI, M.S.; VIENS, J.; MESSADDEQ, Y. PRACTICAL APPLICATION OF ELECTROCHEMICAL NITRATE SENSOR UNDER LABORATORY AND FOREST NURSERY CONDITIONS. Sensors, v. 16, 1190, 2016.
CHETELAT, R.T.; PERTUZÉ, R.A.; FAÚNDEZ, L.; GRAHAM, E.B.; JONES, C.M. DISTRIBUTION, ECOLOGY AND REPRODUCTIVE BIOLOGY OF WILD TOMATOES AND RELATED NIGHTSHADES FORM THE ATACAMA DESERT REGION OF NORTHERN. Euphytica, Chile, v. 167, n. 1, pp. 77-93, 2009.
EMBRAPA HORTALIÇAS. Sistemas de produção, 1 – 2ª Edição. Versão eletrônica. Dez/2006. Acesso em: <https://sistemasdeproducao.cnptia.embrapa.br/FontesHTML/Tomate/TomateIndustrial_2ed/>.
GUIMARÃES, A.C.; MALACHIAS, M.V.B.; COELHO, O.R.; ZILLI, E.C.; LUNA, R.L. USO DO SILDENAFIL EM PACIENTES COM DOENÇA CARDIOVASCULAR. Arquivo Brasileiro de Cardiologia, v.73, n.6, 1999.
KAIWEN, P.; GONG, P.; WANG, J.; WANG, Y.; LIU, C.; LI, W.; ZHANG, L. APPLICATIONS OF NITRATE AND AMMONIUM FERTILIZERS ALTER SOIL NEMATODE FOOD WEBS IN A CONTINUOUS CUCUMBER CROPPING SYSTEM IN SOUTHWESTERN SICHUAN, CHINA. Eurasian Journal of Soil Science, v.4, n.4, pp. 287 – 300, 2015.
LASKOWSKI, R.A.; MACARTHUR, M.W.; MOSS, D.S.; THORNTON,J.M.PROCHECK: A PROGRAM TO CHECK THE STEREOCHEMICAL QUALITY OF PROTEIN STRUCTURES. J. Appl. Cryst., v.26, pp. 283-291, 1993.
































