Detecção de transcrito codificante da histona acetiltransferase (HAT) em Melipona scutellaris
ISBN 978-85-85905-21-7
Área
Bioquímica e Biotecnologia
Autores
Guedes, P.H.G. (UFU) ; Ueira-vieira, C. (UFU) ; Mendes-silva, A.P. (UFMG) ; Fujimura, P.T. (UFU) ; Borges, L.D.F. (UFU) ; Moço, A.C.R. (UFU) ; Bonetti, A.M. (UFU)
Resumo
Em abelhas do gênero Melipona os mecanismos de desenvolvimento e diferenciação de castas não são explicados pela alimentação diferencial das larvas, sendo necessário um componente epigenético. O presente trabalho teve como objetivo detectar o transcrito codificante da histona acetiltransferase (HAT) em Melipona scutellaris. Foi realizada a extração do RNA total de abelhas M. scutellaris nos estágios de larva, pupa e forrageira. A partir do RNA total extraído de cada um dos estágios foi sintetizado o cDNA do transcrito codificante de HAT e RT-PCR foi executada. Os resultados evidenciaram os transcritos de HAT em todos os estágios analisados. Em M. scutellaris, eventos epigenéticos apresentam potencial para o controle de expressão de genes importantes para desenvolvimento dessa abelha.
Palavras chaves
Histona acetiltransferase; Melipona scutellaris; Acetilação de histonas
Introdução
Melipona scutellaris, abelha sem ferrão, popularmente conhecida como Uruçu do Nordeste, Uruçu Verdadeira e Uruçu do Campo, constitui o objeto de estudo do presente trabalho. No Brasil, M. scutellaris ocorre nos estados do Rio Grande do Norte, Sergipe, Ceará, Alagoas, Paraíba, Pernambuco e Bahia (CAMARGO; PEDRO, 2008). Abelhas do gênero Melipona contribuem para aumentar a diversidade genética das plantas por meio de fecundação cruzada, garantindo o sucesso reprodutivo de espécies arbóreas com flores (LOLI, 2008). Além disso, esta espécie de abelhas sem ferrão contribui para renda na agricultura familiar por ser cultivada para produção de mel e própolis (KERR; CARVALHO; NASCIMENTO, 1996; VELIKOVA et al., 2000; DE SOUSA et al., 2013). A determinação de castas para a maioria dos Hymenoptera é via alimentação diferencial da larva (KERR; NIELSEN, 1966). Em abelhas Apis mellifera, as larvas se tornam rainhas ou operárias dependendo da quantidade do alimento recebido durante o estágio larval (BEETSMA, 1979). No gênero Melipona, o processo de alimentação das larvas é massivo e não foi observada diferença qualitativa em relação ao alimento destinado às larvas que originarão rainhas e operárias (KERR, 1950). Em função disso, Kerr (1948; 1950) propôs o mecanismo genético-alimentar para determinação de casta em Melipona. Contudo, mostram-se insuficientes para explicar os mecanismos de desenvolvimento e diferenciação de castas em M. scutellaris. Como alternativa, eventos epigenéticos podem esclarecer essas questões ainda não completamente elucidadas. Acetilação, fosforilação, metilação de histonas e metilação das citosinas do DNA são os marcadores epigenéticos mais estudados (KOUZARIDES, 2007). Sem alterar a sequência de nucleotídeos do genoma, tais mecanismos promovem padrões globais epigenéticos hereditários denominados epigenoma (PATALANO et al., 2012). A acetilação de histonas torna neutra a carga positiva das cadeias laterais dos resíduos o que enfraquece a interação eletrostática da histona com a molécula de DNA, acarretando no relaxamento da estrutura da cromatina e permitindo acesso de proteínas regulatórias aos promotores dos genes (TURNER, 2000). O presente trabalho buscou detectar transcritos do gene codificador da enzima histona acetiltransferase (HAT), responsável por regular a expressão gênica via remodelagem da cromatina nos estágios de larva (terceiro estágio, terceiro instar), pupa de corpo branco e olho rosa de operária (PpW) e forrageira (F) de Melipona scutellaris.
Material e métodos
Abelhas sem ferrão da espécie Melipona scutellaris nos estágios de larva de L3-3 (larva do terceiro estágio, terceiro instar); estágio de pupa de corpo branco e olho rosa de operária (PpW); e abelha na fase adulta forrageira (F). A extração de RNA total foi executada pelo método do TRIZOL (solução comercial de fenol e isotiocianato de guanidina para isolamento de RNA total, Ambion – Life Technologies), seguindo as recomendações do fabricante. O RNA foi quantificado em Nanodrop (ND-1000 Spectrophotometer) a 260 nm. Para a síntese de cDNA (RT) foi utilizada a enzima M-MLV transcriptase reverse (Promega) e Oligo dT (15) (Invitrogen), seguindo as instruções dos fabricantes. A segunda fita foi sintetizada por PCR com o uso de primers específicos para os genes alvo. A qualidade do cDNA foi testada, com transcrito do gene codificador da proteína ribossomal 49, rp49 (GenBank accession number AF441189) que além de um fragmento de 150 pb, amplifica um de 240 pb no caso de contaminação com DNA genômico (LOURENÇO et al., 2008). Os primers utilizados foram desenhados utilizando o programa Oligo Explorer (Version 1.2). Os primers foram desenhados com base nas sequências encontradas por homologia no genoma de M. quadrifasciata, uma vez que esse organismo apresenta maior homologia com as abelhas M. scutellaris. Por fim, a migração eletroforética foi realizada em gel de agarose 1,5% acompanhada de marcador de peso molecular para avaliação do tamanho do fragmento obtido.
Resultado e discussão
A Figura 1 apresenta a amplificação, por meio de RT-PCR, do transcrito codificante de HAT,
seguido de eletroforese em gel de agarose 1,5% corado com brometo de etídeo, e o marcador
utilizado foi de 50pb (M50pb). CN na Figura 1 é controle negativo do experimento. Com base neste
resultado, verificou-se que todas as amostras analisadas possuem a presença do transcrito
codificador de HAT (banda de 79pb indicado com seta branca) envolvido na maquinaria
epigenética de acetilação de histonas. A acetilação de histonas modula a transcrição gênica de
múltiplas formas. A acetilação de grupos ε-amino de resíduos de lisina nas caudas das histonas
neutraliza a carga positiva, relaxando assim a estrutura da cromatina. Isso interfere com as
estruturas da cromatina e aumenta a acessibilidade dos fatores de transcrição para os promotores
de genes alvo. As histonas acetiladas também servem como locais de ligação para as proteínas de
bromodomínio que, geralmente, agem como ativadores da transcrição (HABERLAND et al., 2009).
Resultados preliminares do transcriptoma de corpora allata de larvas (UEIRA-VIEIRA et al., em
preparação) comprovam presença de genes que codificam enzimas relacionadas com a
maquinaria epigenética em M. scutellaris. Além disso, diferenças na dispersão e volume da
heterocromatina em rainhas e operárias também já foram identificadas em Melipona scutellaris
(BORGES, 2014). Nesse sentido, os resultados do nosso estudo indicam que a presença de
transcritos codificantes da enzima HAT corroboram com resultados descritos na literatura
anteriormente e podem estar envolvidos na transição de estágios em abelhas sem ferrão Melipona
scutellaris.
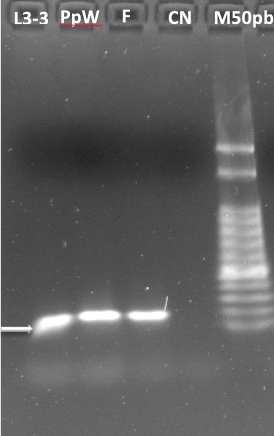
Eletroforese da amplificação do transcrito de HAT por RT-PCR em M. scutellaris. A seta branca indica banda (79pb) de amplificação do transcrito.
Conclusões
Abelhas sem ferrão são excelentes modelos biológicos para pesquisas com ênfase em eventos epigenéticos, neste caso a acetilação de histonas, associados a transição de estágios em abelhas sem ferrão. Além disso, os primers específicos desenhados para a identificação de transcritos do gene codificante da enzima HAT apresentaram boa amplificação nas amostras analisadas. Finalmente, foi possível, pela primeira vez em abelhas Melipona scutellaris, identificar transcritos relacionados à maquinaria epigenética de acetilação de histonas.
Agradecimentos
CAPES, CNPQ, FAPEMIG e LABGEN UFU.
Referências
BEETSMA, J. The process of queen-worker differentiation in the honeybee. Bee World. v. 60, p. 24-39, 1979.
BORGES, N. A. Padrão de Heterocromatina em corpora allata de Melipona scutellaris (Apidae, Meliponini).2014. Dissertação (Mestrado) - Programa de Pós-graduação em Genética e Bioquímica, Universidade Federal de Uberlândia, Uberlândia, 2014.
CAMARGO, J.M.F.; PEDRO, S.R.M. Revisão das espécies de Melipona do grupo fuliginosa (Hymenoptera, Apoidea, Apidae, Meliponini). Rev. Bras. p. 411-427, 2008.
DE SOUSA, C. S. et al. Insights into the Melipona scutellaris (Hymenoptera, Apidae, Meliponini) fat body transcriptome. Genetics and Molecular Biology, v. 36, n. 2, p. 292– 297, 2013.
HABERLAND, M. et al. The many roles of histone deacetylases in development and physiology: implications for disease and therapy. Nature Reviews Genetics, v. 10, p. 32–42, 2009.
KERR, W. E. Estudos sobre o Gênero Melipona. Anais da Escola Superior de Agronomia “Luiz de Queiroz”, v. 5, p. 181–276, 1948.
KERR, W. E. Genetic determination of castes in the genus Melipona. Genetics, v. 35, n.
2, p. 143, 1950.
KERR, W.E.; NIELSEN, R.A. Evidences that genetically determined melipona queens can become workers. Genetics, p. 859-866, 1966.
KERR, W. E.; CARVALHO, G. A.; NASCIMENTO, V. A. Abelha Urucu: Biologia, Manejo e Conservação. Belo Horizonte: Editora Liber Liber, 1996.
KOUZARIDES, T. Chromatin modifications and their function. Cell. v. 128, n.4, p. 693- 705, fev. 2007
LOLI, D. Termorregulação colonial e energética individual em abelhas sem ferrão Melipona quadrifasciata Lepeletier (Hymenoptera, Apidae, Meliponini). p. 229, 2008.
LOURENÇO, A. P. et al. Validation of reference genes for gene expression studies in the honey bee, Apis mellifera , by quantitative real-time RT-PCR. Apidologie, v. 39, n. 3, p.372- 385, 2008.
PATALANO, S. et al. Shifting behaviour: Epigenetic reprogramming in eusocial insects.Current Opinion in Cell Biology, v. 24, n. 3, p. 367–373, 2012.
TURNER, B. M. Histone acetylation and an epigenetic code. BioEssays, v. 22, n. 9, p.836–845, 2000.
VELIKOVA, M. et al. Chemical composition and biological activity of propolis from Brazilian Meliponinae. Zeitschrift fur Naturforschung - Section C Journal of Biosciences, v. 55, n. 9-10, p. 785–789, 2000.
































