ÁREA: Produtos Naturais
TÍTULO: Sítio de ligação do oleato à proteína FAR-7 de Caenorhabditis elegans
AUTORES: Oliveira, D. (UFLA) ; Campos, V. (UFLA)
RESUMO: A proteína FAR7 de Caenorhabditis elegans foi computacionalmente corrigida e
submetida a processo de dinâmica molecular, o que permitiu selecionar conformações
para a realização de ancoragem do dec-1-eno e do octanoato. As regiões preferidas
para ancoragem destas substâncias foram empregadas para a ancoragem do oleato
[(Z)-octadec-9-enoato]. Os complexos FAR7-oleato de menores energias foram
submetidos a processo de dinâmica molecular, o que permitiu observar três
possibilidades com energias livres de ligação em torno de 7,5 kcal/mol. Em todas
as possibilidades o grupo alquila terminal do oleato (C11-C18) ficou próximo dos
resíduos de aminoácidos 10, 14, 92, 93, 96, 105, 106, 110 e 111 da proteína,
enquanto o carboxilato ocupou diferentes posições no complexo.
PALAVRAS CHAVES: Nematoide; ácido graxo; docking
INTRODUÇÃO: Os nematoides causam grandes prejuízos aos agricultores em geral (DIAS-ARIEIRA
et al., 2010). Em decorrência, faz-se necessário desenvolver novos métodos para
o controle desses fitoparasitas. Para tanto, uma das possibilidades diz respeito
à utilização de produtos que possam complexar-se a fatty acid and retinol
binding protein (FAR), já que a mesma é utilizada pelo nematoide para complexar
ácidos graxos insaturados produzidos pelas plantas, o que interrompe parte do
mecanismo de defesa das mesmas contra os nematoides (DAVIS et al., 2008; WUBBEN
et al., 2010). Como ainda não foram identificadas as estruturas tridimensionais
de proteínas FAR produzidas por nematoides fitoparasitas, iniciou-se o trabalho
com a enzima FAR-7 de Caenorhabditis elegans, que é um nematoide de vida livre.
Tal proteína está depositada no Protein Data Bank ((http://www.rcsb.org) sob o
código 2W9Y. Buscou-se no presente estudo identificar, através de métodos
computacionais, o sítio de complexação do oleato à proteína FAR-7 de C. elegans,
para utilizar tal informação no desenvolvimento futuro de novos produtos para o
controle de nematoides fitoparasitas.
MATERIAL E MÉTODOS: Com o programa SPDBV 4.0.2, fizeram-se correções na proteína FAR-7. As 21
conformações resultantes foram submetidas à otimização por cálculos de mecânica
molecular no vácuo e, em seguida, foram alinhadas com o programa VMD 1.9. As
três melhores estruturas obtidas foram colocadas em caixas de água e submetidas
a processo de dinâmica molecular por 400 ps, com intervalos de 2 fs, em
condições de fronteira periódica, com temperatura e pressão constantes. Para
tanto, empregou-se o programa NAMD 2.8 com campo de força do CHARMM. As três
conformações de menores energias em cada processo foram comparadas pelo emprego
dos métodos DFIRE2, QMEAN e VADAR. A estrutura que se mostrou a mais estável foi
submetida a processo de dinâmica molecular por mais 200 ps nas mesmas condições.
Três conformações de menores energias foram selecionadas do último processo de
dinâmica molecular e empregadas em ancoramento as cegas com octanoato e dec-1-
eno, empregando-se os programas AUTODOCK 4.2, AUTODOCK VINA 1.1.2 e IGEMDOCK
2.1. As regiões ao redor dos locais dos ancoramentos, que correspondiam as
melhores poses de cada um dos programas, foram utilizadas para o ancoramento do
oleato [(Z)-octadec-9-enoato] com os mesmos softwares. Em seguida, os complexos
proteína-oleato contendo as melhores poses de cada software de ancoramento foram
submetidos a processo de dinâmica molecular por 300 ps, conforme descrito acima.
Os complexos FAR7-oleato de menores energias em cada um dos processos foram
submetidos ao método BAPPL para cálculo da energia livre de ligação do oleato
(∆Go). Os três complexos de menores energias foram submetidos ao processo de
dinâmica molecular por mais 4 ns e, ao final, as energias livres da ligação do
oleato à proteína foram calculados por MM-PBSA, utilizando o programa SIETRAJ.
RESULTADOS E DISCUSSÃO: As correções iniciais da proteína foram necessárias, já que a estrutura do PDB
ainda continha a tag, havia uma ligação O-CYS9 e o loop referente aos resíduos
42-45 não estava presente. Após este processo se observou a formação de uma
ligação S-S entre os resíduos CYS42 e CYS98. Inicialmente se descartou uma das
estruturas de cada par cujo desvio quadrático médio (RMSD) era menor ou igual a
0,05 Ǻ. Estruturas que se chocavam com três ou mais moléculas de água de
cristalização de FAR7 também foram descartadas. Como o ácido oleico apresentava
grande número de conformações, optou-se por dividi-lo em duas estruturas na
parte inicial do processo de ancoramento: octanoato e dec-1-eno. Desta forma,
não foi necessário fazer ancoramento as cegas com o oleato, o que reduziu
bastante a complexidade do problema. Os complexos FAR7-oleato de menores
energias após a primeira etapa de dinâmica molecular foram submetidos ao método
BAPPL para cálculo da energia livre de ligação do oleato (∆Go). Os valores mais
favoráveis ficaram entre -7,63 e -7,42 kcal/mol. Após 4 ns de dinâmica molecular
os três (Figura 1) complexos inicialmente selecionados continuaram a apresentar
valores de energias livres de ligação próximos, em torno de 7,5 kcal/mol, o que
sugere que em equilíbrio as três possibilidades devam coexistir.
Complexo FAR7 e oleato
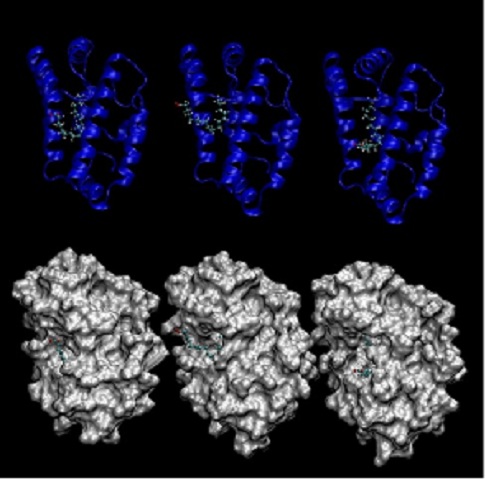
A figura apresenta os três melhores resultados para
o complexo FAR7 e oleato após dinâmica molecular.
CONCLUSÕES: O presente estudo demonstrou, pelo emprego de métodos in silico, que o oleato se
complexa à FAR7 de C. elegans de forma que os grupos alquilas terminais (C11-C18)
estejam próximos dos resíduos resíduos de aminoácidos 10, 14, 92, 93, 96, 105,
106, 110 e 111 da proteína FAR7. Quanto ao grupo carboxilato, aparentemente tem
maior liberdade de movimentação. Com isto, o sítio de complexação foi
identificado, o que permitirá desenvolver produtos com maiores afinidades por tal
sítio que ácidos graxos em geral.
AGRADECIMENTOS: CAPES, CNPq, FAPEMIG e CESUP-UFRGS, onde parte do trabalho foi desenvolvido.
REFERÊNCIAS BIBLIOGRÁFICA: DAVIS, E.L.; HUSSEY, R.S.; BAUM, T.J. Parasitism Genes: What They Reveal about Parasitism. In: R. Howard Berg and Christopher G. Taylor (Eds), Cell Biology of Plant Nematode Parasitism, Springer, p. 14-44, 2008.
DIAS-ARIEIRA, C. R., FURLANETTO, C., SANTANA, S. M., BARIZÃO, D. A. O., RIBEIRO, R. C. F. (2010). Fitonematoides associados a frutíferas na região noroeste do Paraná, Brasil. Rev. Bras. Frutic., 32, 1064-1071.
WUBBEN, M.J.; CALLAHAN, F.E.; SCHEFFLER, B.S. Transcript analysis of parasitic females of the sedentary semi-endoparasitic nematode Rotylenchulus reniformis. Molecular & Biochemical Parasitology 172, 31–40, 2010.
